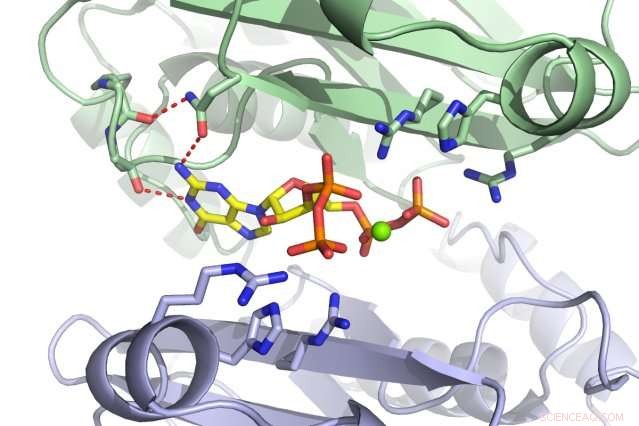
Uma equipe do MIT descobriu que a pequena molécula ppGpp (vermelha) se liga à enzima PurF (subunidades mostradas em verde e roxo). Crédito:Massachusetts Institute of Technology
Em 1969, O cientista Michael Cashel estava analisando os compostos produzidos por bactérias famintas quando notou dois pontos aparecendo em seu cromatograma como por mágica. Hoje, nós conhecemos um desses "pontos mágicos, "como os pesquisadores os chamam, como tetrafosfato de guanosina, ou ppGpp para breve. Também entendemos que é uma molécula sinalizadora presente em praticamente todas as bactérias, ajudando a ajustar o crescimento e o tamanho das células com base na disponibilidade de nutrientes.
E ainda, apesar de décadas de estudo, exatamente como o ppGpp regula o crescimento bacteriano permanece um tanto misterioso. Investigar ainda mais requer uma lista mais abrangente das moléculas que o ppGpp se liga para exercer seus efeitos.
Agora, colaboradores dos departamentos de Biologia e Química do MIT desenvolveram um método para fazer exatamente isso, e usaram sua nova abordagem para localizar mais de 50 alvos ppGpp em Escherichia coli - quase a metade dos quais não haviam sido identificados anteriormente. Muitos desses alvos são enzimas necessárias para produzir nucleotídeos, os blocos de construção de DNA e RNA. Durante os momentos em que as bactérias não têm nutrientes suficientes para crescer e se dividir normalmente, os pesquisadores propõem que o ppGpp impede que essas enzimas criem novos nucleotídeos do zero, ajudando as células a entrar em um estado dormente.
"Com pequenas moléculas ou metabólitos como ppGpp, tem sido difícil, historicamente, determinar quais proteínas elas se ligam, "diz Michael Laub, um professor de biologia, um investigador do Howard Hughes Medical Institute, e o autor sênior do estudo. "Este tem sido um problema intratável que atrasou o campo por algum tempo, mas nossa nova abordagem permite que você identifique os alvos prováveis em questão de semanas. "
O pós-doutorado Boyuan Wang é o primeiro autor do estudo, que aparece em Nature Chemical Biology em 17 de dezembro.
O local mágico
Como o ppGpp foi descoberto há quase 50 anos, foi demonstrado que suprime a replicação do DNA, transcrição, tradução, e várias vias metabólicas. Isso freia o crescimento celular e permite que as bactérias persistam em face da fome, estresse, e antibióticos. Sua influência sobre vários processos regulatórios permaneceu um tanto misteriosa, no entanto, afinal, ele não apenas modula um único caminho, mas coordena várias operações simultaneamente para orquestrar um desligamento em massa da célula.
A fim de discernir quais proteínas o ppGpp se liga para efetuar essa mudança generalizada, os pesquisadores construíram o que eles chamam de "compostos de captura" que contêm ppGpp, permitindo-lhes pescar seus alvos a partir de extratos bacterianos. Esses compostos incluíram um reticulador fotorreativo que se fixou firmemente nas proteínas de interesse na presença de luz, e um identificador de biotina que ajudou os cientistas a retirar as proteínas para identificá-las. Mais importante, eles foram unidos ao ppGpp de forma que não interferissem em sua capacidade de vincular-se a seus alvos. Este método é mais eficiente e preciso em comparação com os meios mais tradicionais de distinguir alvos ppGpp, que são muito mais árduas e carecem de sensibilidade.
"Nossa abordagem resolve esses problemas porque você não é mais obrigado a fazer esses protocolos de mão-de-obra intensiva para identificar alvos ppGpp - e funciona até mesmo em bactérias além da E. coli, "diz Wang." Embora o ppGpp seja comum entre muitas espécies bacterianas, parece exercer seus efeitos por meio de diferentes mecanismos, o que complica as coisas. Nossos compostos de captura fornecem uma maneira de desvendar essa diversidade, e em pouco tempo. "
Pondo um freio na produção de nucleotídeos
Embora o 56 ppGpp tenha como alvo Wang identificou em sua tela o controle de uma miríade de processos celulares, ele se concentrou na enzima PurF - que inicia a biossíntese de nucleotídeos de purina contendo bases de adenina e guanina, também conhecido como A e G.
Quando as bactérias estão estressadas ou morrem de fome, eles entram em um estado dormente para sobreviver. Mas simplesmente restringir a tradução e a transcrição não é suficiente; os nucleotídeos ainda estão sendo gerados e irão se acumular se sua síntese não for colocada em pausa. As células podem construir nucleotídeos de duas maneiras:resgatando materiais existentes ou começando do zero. PurF dá início à primeira etapa do último processo, levando aos nucleotídeos A e G. Contudo, quando ppGpp se liga a PurF, faz com que a enzima mude de forma, o que o impede de fazer seu trabalho, reduzindo assim a produção de nucleotídeos na célula.
"Esta é a primeira vez que uma enzima envolvida nessa via ou função específica foi identificada como um alvo ppGpp, "Diz Wang." Se você limitar o consumo de nucleotídeos, mas não sua produção, o pool de nucleotídeos vai explodir, o que não é bom para o celular. Então, mostramos que o ppGpp também resolve esse problema. "
Além de PurF e outras enzimas necessárias para a produção de nucleotídeos, os pesquisadores notaram que o ppGpp também se liga a muitas enzimas GTPase envolvidas na tradução. Isso pode indicar um mecanismo à prova de falhas que retarda a tradução ao atingir vários, enzimas semelhantes de uma maneira quase redundante em face da fome.
Enquanto Wang continua a refinar seu método, ele visa aumentar sua especificidade e garantir que seus compostos de captura se liguem às mesmas proteínas que fariam dentro de uma célula viva. Ele também espera rastrear proteínas de ligação ppGpp em outras bactérias, incluindo patógenos que dependem do ppGpp para sobreviver dentro de seus hospedeiros e propagar doenças como a tuberculose.
"Esta é uma abordagem química empolgante para entender melhor a função de uma molécula de sinalização conservada há muito estudada nas bactérias, "diz Jue Wang, professor de bacteriologia da Universidade de Wisconsin em Madison, que não estava envolvido com o estudo. "Suas descobertas e técnicas são altamente relevantes para muitas outras bactérias, e vai melhorar muito o conhecimento de como as bactérias usam essa molécula de sinalização crítica para mediar tudo, desde a sobrevivência no intestino humano até causar doenças. "
Laub acrescenta:"Ainda hoje estamos descobrindo novas moléculas de sinalização baseadas em nucleotídeos em bactérias, e cada um deles poderia eventualmente ser derivatizado de forma semelhante para identificar seus parceiros de ligação. "
Esta história foi republicada por cortesia do MIT News (web.mit.edu/newsoffice/), um site popular que cobre notícias sobre pesquisas do MIT, inovação e ensino.