Bibliotecas combinatórias são um componente chave do kit de ferramentas do químico para a triagem de ligantes. Bibliotecas combinatórias dinâmicas adicionam uma nova dimensão ao interligar síntese e triagem. Agora, Cientistas britânicos desenvolveram uma biblioteca combinatória dinâmica para a triagem de estruturas de ligantes supramoleculares que reconhecem superfícies de proteínas e podem interferir na interação proteína-proteína. O estudo foi publicado no European Journal of Organic Chemistry .
A química combinatória foi adotada pela indústria farmacêutica porque permite aos químicos criar grandes quantidades de moléculas e testá-las quanto às propriedades desejadas, por exemplo, afinidades de ligação a um alvo terapeuticamente importante. A síntese e a triagem estão ligadas na química combinatória dinâmica, pois as bibliotecas combinatórias dinâmicas podem mudar sua distribuição de produto em resposta à ligação ao alvo. Este processo de amplificação do produto gera estruturas supramoleculares que são de difícil acesso e permite a identificação rápida de arquiteturas de ligação, por exemplo, inibidores de ligação ao sítio ativo de uma enzima.
Contudo, superfícies de proteínas são grandes estruturas tridimensionais com manchas de hidrofobicidade diferente, acidez, e basicidade, encontrados em posições díspares em sua superfície. Portanto, a triagem de inibidores de interações proteína-proteína deve envolver ligantes mais complexos do que aqueles necessários para o reconhecimento do sítio ativo "Precisávamos identificar química reversível e metodologia analítica que seria compatível com a ideia de fazer química combinatória dinâmica para reconhecimento de superfície de proteína, "diz Andrew J. Wilson, o autor correspondente do estudo que foi realizado na Universidade de Leeds.
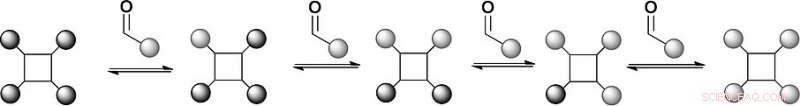
Os cientistas escolheram uma estrutura de tetrafenilporfirina contendo quatro hidrazidas e benzaldeídos substituídos como braços de reconhecimento que se ligariam reversivelmente às hidrazidas para formar hidrazonas. As porfirinas são estruturas estabelecidas para o reconhecimento da superfície da proteína, mas a química da hidrazona é um desafio sintético. "Temos um andaime com quatro hidrazonas para que possamos fazer troca química em quatro posições diferentes, "diz Wilson. Além disso, química de formação de hidrazona reversível geralmente ocorre em um pH ácido, mas as proteínas preferem um pH neutro. Os cientistas resolveram esse problema adicionando um catalisador à base de anilina, que empurrou a reação de troca de hidrazona em direção ao equilíbrio termodinâmico em pH próximo ao neutro.
Na primeira configuração desta biblioteca de modelos, os pesquisadores tentaram dois ligantes de aldeído substituídos, que formou um equilíbrio com a hidrazona derivada de benzaldeído. A análise de espectrometria de massa revelou que a distribuição do produto estava em equilíbrio termodinâmico, e os ligantes de fato trocados por reação reversível. Isso significa que as condições para uma biblioteca combinatória dinâmica para reconhecimento de superfície de proteína foram definidas. Próximo, os cientistas planejam elaborar esta abordagem para estudar a seleção dirigida por proteínas de miméticos de superfície de proteínas.