
Hiroaki Kiyokawa, MD, PhD, professor de Farmacologia e Patologia, foi co-autor sênior em estudos publicados em Nature Communications e Avanços da Ciência . Crédito:Northwestern University
Os investigadores da Northwestern Medicine desenvolveram uma técnica para catalogar como as células descartam proteínas desnecessárias, um processo que tem implicações para o câncer e doenças do espectro do autismo, de acordo com as descobertas publicadas em Nature Communications .
Defeitos na ubiquitinação, ou como as células descartam proteínas desnecessárias, têm sido implicados em uma variedade de doenças, mas os cientistas não tinham anteriormente um método confiável para rastrear as enzimas envolvidas neste processo de descarte, de acordo com Hiroaki Kiyokawa, MD, Ph.D., professor de Farmacologia e co-autor sênior do estudo.
Para combater isso, uma equipe colaborativa liderada por Kiyokawa e Jun Yin, Ph.D., professor associado de química na Georgia State University, desenvolveram transferência ortogonal de ubiquitina, um método de rastrear quais enzimas desencadeiam o descarte de quais proteínas. Esta técnica pode dar aos cientistas uma melhor compreensão dos mecanismos de doenças decorrentes da ubiquitinação anormal, esperançosamente levando a uma terapia melhor, Kiyokawa disse.
Ubiquitinação sinaliza proteínas para destruição e é facilitada por três classes de enzimas; E1, Enzimas E2 e E3. Ubiquitina é ligada às três enzimas em sequência, mas as enzimas E1 e E2 são enzimas iniciadoras simples, portanto, as células de mamíferos têm apenas um número limitado de variedades E1 e E2. Por outro lado, existem centenas de enzimas E3, sugerindo que cada E3 tem seu próprio alvo de proteína único.
"A relação entre as enzimas E3 e a proteína alvo é como uma fechadura e uma chave, "disse Kiyokawa, também professor de Patologia e membro do Robert. H. Lurie Comprehensive Cancer Center da Northwestern University.
A diversidade de proteínas-alvo e as interações fracas significam que pode ser um desafio identificar claramente qual enzima E3 marca qual proteína. Contudo, a transferência ortogonal de ubiquitina (OUT) é uma nova solução para o problema, de acordo com Kiyokawa.
Em OUT, cientistas desenvolveram uma via de ubiquitina artificial que imita o sistema nativo, mas responde apenas à ubiquitina mutante, que tem uma etiqueta especial que permite aos cientistas identificar a proteína que ele eventualmente almeja no final do ciclo.
“Existem duas vias dentro das mesmas células, "Kiyokawa disse." O original é como uma rodovia, e o caminho planejado é um desvio. "
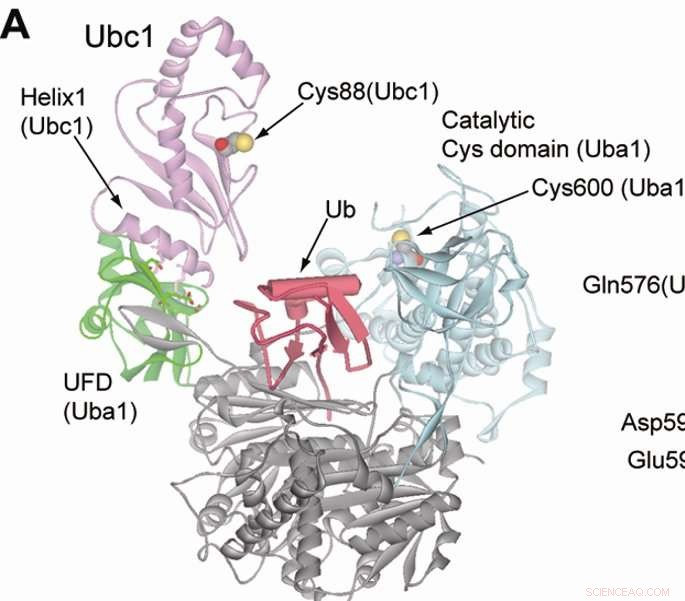
Ilustração que mostra a ubiquitina passando de uma enzima E1 para uma enzima E2. Crédito:Northwestern University
No estudo atual, os investigadores construíram uma via artificial para uma enzima E3 chamada E6AP, e introduziu a ubiquitina mutada e as vias nas células. Depois de uma tela de proteômica, os cientistas foram capazes de identificar os alvos proteicos específicos do E6AP.
"Este estudo é apenas uma vitrine da tecnologia, mas acreditamos que podemos aplicar isso a muitas das 600 proteínas E3 sem muitos problemas, "Kiyokawa disse." Muitas enzimas E3 são conhecidas por serem superexpressas ou mutadas em doenças neuronais, portanto, há um grande conjunto de oportunidades de pesquisa. "
Contudo, E6AP, em particular, demonstrou ter como alvo proteínas supressoras de tumor quando ativadas de forma inadequada pelo papilomavírus humano, contribuindo para o aumento do risco de câncer enfrentado por pacientes infectados. Além disso, o locus do gene que codifica para E6AP é conhecido por ser mutado na síndrome de Angelman, um transtorno do espectro do autismo.
"Ainda não sabemos por que o E6AP precisa ser mutado para mostrar os sintomas da síndrome de Angelman, mas a investigação de alvos específicos pode nos ensinar mais sobre os mecanismos fisiopatológicos da doença autista, "Kiyokawa disse.
Outras investigações sobre alvos específicos já estão em andamento — Kiyokawa e colegas publicaram recentemente outro estudo envolvendo OUT, desta vez em Avanços da Ciência .
Eles encontraram uma enzima E3 diferente, LASCA, estava envolvido na degradação de CDK4, uma enzima que desempenha um papel fundamental na proliferação celular.
"Parece ser um mecanismo que pode interromper o ciclo celular sob estresse, "Kiyokawa disse." Se este regulamento for quebrado, o organismo pode ser mais suscetível ao câncer. "
Ainda, não é suficiente simplesmente identificar esses alvos de ubiquitinação, de acordo com Kiyokawa.
"Precisamos aprender por que esses alvos recém-definidos estão sob a regulação das enzimas E3 se quisermos encontrar novos alvos terapêuticos, "disse ele." Só então poderemos desenvolver medicamentos ou tratamentos. "