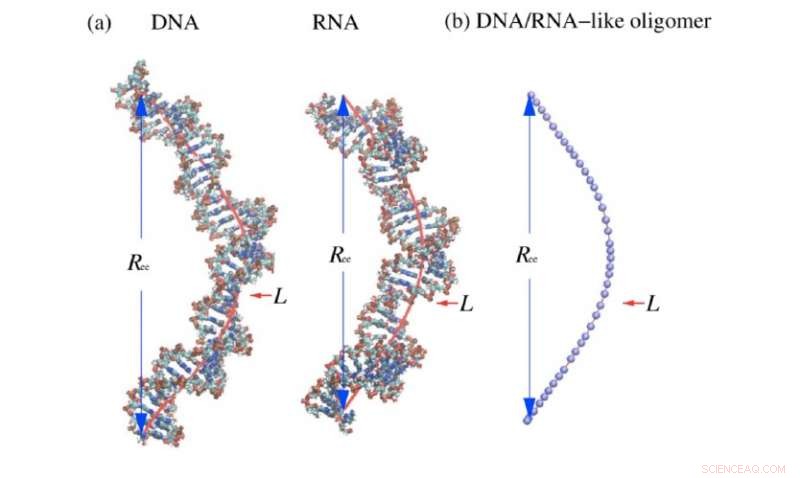
Ilustrações de DNA de fita dupla, RNA e um modelo de cadeia de contas semelhante a um verme. A distribuição radial da distância de ponta a ponta, Ree, e comprimento do contorno, EU, são mostrados. Crédito:Xi Zhang / Bao / Wu / Zhu / Tan
Ao contrário dos modelos de plástico rígido da aula de química, cadeias reais de moléculas podem dobrar e esticar, como contas em um cordão elástico. Alguns polímeros, como DNA, são especialmente elásticos, uma característica que pode complicar as tentativas de modelar seu comportamento.
Desde o trabalho seminal de Paul Flory, os pesquisadores desenvolveram uma variedade de fórmulas para calcular a distância entre as extremidades de um polímero curvo. Contudo, essas fórmulas normalmente não consideram a elasticidade da molécula. Em um novo estudo, publicado esta semana em The Journal of Chemical Physics , cientistas desenvolveram uma fórmula para determinar a distância de ponta a ponta de um polímero semiflexível, incluindo DNA ou RNA, levando em consideração o quanto o polímero se estica.
Estimativas anteriores de como os polímeros se dobram não levaram em conta como a molécula se move em três dimensões. "Este método para calcular a distribuição do comprimento do contorno é mais rigoroso, "disse Xi Zhang da Wuhan University e principal autor do artigo." Não só podemos calcular a distância de ponta a ponta, também podemos descobrir a forma do polímero. "
Ao incluir a elasticidade do polímero, a nova fórmula pode ajudar os pesquisadores a estimar a flexibilidade dos segmentos de DNA, uma propriedade sabidamente essencial para sua função biológica. A flexibilidade do DNA impacta a ligação de proteínas regulatórias e como o DNA envolve as histonas, proteínas que agem como carretéis para manter o DNA perfeitamente empacotado dentro de um núcleo. As formas específicas que o DNA se curva e envolve em torno das histonas podem afetar a expressão do gene, expondo certos genes para o exterior, enquanto outros permanecem escondidos.
Os pesquisadores construíram a base do modelo de cadeia semelhante a um verme, que trata polímeros semiflexíveis como DNA e RNA como links em uma cadeia. Usando extensas simulações de Monte Carlo, eles validaram sua fórmula em uma ampla gama de valores de elasticidade e flexibilidade. Eles também usaram simulações de dinâmica molecular, qual modelo como as moléculas se movem e interagem no tempo, para garantir que eles obtiveram resultados semelhantes de seu método para polímeros curtos de DNA e RNA.
Este tipo de fórmula é mais eficiente computacionalmente do que usar simulações de computador para determinar a distância de ponta a ponta do alongamento, polímeros de dobra, e, em segundos, pode calcular resultados que podem levar semanas de simulações.
A nova fórmula é especialmente útil para estimar a distribuição de comprimento de ponta a ponta de pequenos polímeros, apontam os autores. "Esse alongamento é muito importante em um biopolímero quando ele é muito curto, digamos 40 pares de bases, "Zhang disse. Eles calculam que o efeito do alongamento se torna insignificante para moléculas de DNA maiores do que cerca de 130 pares de bases, e para RNAs mais longos do que cerca de 240 pares de bases.