A rotação impulsionada pela hidrólise do ATP da haste central na ATP sintase do tipo V da bactéria Enterococcus hirae descreve o acoplamento quimio-mecânico do motor molecular. Essas simulações foram realizadas no supercomputador Titan usando NAMD2.12. A visualização e a renderização do filme foram concluídas em VMD1.9.3. Crédito:Barry Isralewitz, Universidade de Illinois em Urbana-Champaign
Apesar da grande diversidade entre os organismos vivos, a molécula usada para armazenar e transmitir energia dentro do aeróbio, ou usando oxigênio, células é notavelmente o mesmo. De bactérias a fungos, plantas, e animais, adenosina trifosfato (ATP) serve como a moeda universal de energia da vida, alimentando os processos de que as células precisam para sobreviver e funcionar.
Ao longo de um dia, um indivíduo normalmente usará o equivalente ao seu peso corporal em ATP; Contudo, o corpo humano carrega apenas uma pequena quantidade da molécula por vez. Isso significa que as células devem reciclar ou reabastecer constantemente sua capacidade limitada, contando com um motor molecular altamente eficiente chamado ATP sintase para fazer o trabalho.
Como parte de um projeto dedicado a modelar como bactérias roxas unicelulares transformam luz em alimento, uma equipe de cientistas computacionais da Universidade de Illinois em Urbana-Champaign (UIUC) simulou uma ATP sintase completa em detalhes de todos os átomos. O trabalho se baseia na primeira fase do projeto - uma organela fotossintética de 100 milhões de átomos chamada cromatóforo - e dá aos cientistas um vislumbre sem precedentes de uma máquina biológica cuja eficiência energética ultrapassa em muito a de qualquer sistema artificial.
Proposto pela primeira vez sob a liderança do falecido Klaus Schulten, pioneira no campo da biofísica computacional e fundadora do Grupo de Biofísica Teórica e Computacional da UIUC, a pesquisa progrediu sob a liderança de Abhishek Singharoy, co-investigador principal e bolsista de pós-doutorado da National Science Foundation no Centro de Física das Células Vivas da UIUC.
Além de Singharoy, a equipe inclui membros dos grupos de professores da UIUC Emad Tajkhorshid, Zaida Luthey-Schulten e Aleksei Aksimentiev; o cientista pesquisador Melih Sener; e os desenvolvedores Barry Isralewitz, Jim Phillips, e John Stone. O colaborador experimental Neil Hunter, da University of Sheffield, na Inglaterra, também participou do projeto.
A equipe liderada pela UIUC construiu e testou seu megamodelo sob uma alocação de vários anos concedida por meio do programa Impacto Computacional Inovador e Novo em Teoria e Experimento no supercomputador Titan, um Cray XK7 gerenciado pelo Oak Ridge Leadership Computing Facility (OLCF) do Departamento de Energia dos EUA (DOE), um DOE Office of Science User Facility localizado no Oak Ridge National Laboratory do DOE.
Usando Titan, a equipe produziu uma ferramenta virtual que pode prever com detalhes exatos a produção de energia química de um sistema fotossintético com base na quantidade de luz solar absorvida. A pesquisa pode um dia contribuir para uma tecnologia avançada de energia limpa que incorpore conceitos biológicos.
"A natureza projetou o cromatóforo de forma que ele possa gerar ATPs suficientes para que essas bactérias sobrevivam em ambientes de pouca luz, como o fundo de lagoas e lagos, "Singharoy disse." Nosso trabalho capturou esse processo de conversão de energia em detalhes de todos os átomos e nos permitiu prever sua eficiência. "
Luz em movimento
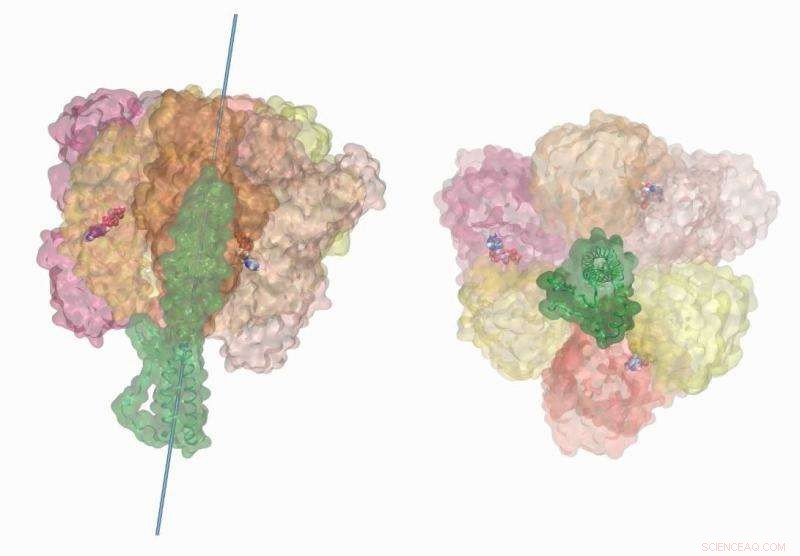
Muitas vezes referida como a usina de energia da célula, A ATP sintase é uma enzima complexa que acelera a síntese de seus precursores moleculares, difosfato de adenosina (ADP) e fosfato. Incorporado na membrana interna e externa do cromatóforo, o motor enzimático consiste em três partes principais - um rotor movido a íons, uma haste central, e um anel de proteína.
Semelhante a uma roda d'água que gira com a força de um riacho, o rotor ATP sintase aproveita o movimento eletroquimicamente estimulado de íons, como prótons ou sódio, de alta concentração a baixa concentração através da membrana. A energia mecânica resultante é transferida para a haste central, que auxilia o anel de proteína na síntese de ATP.
Notavelmente, o processo funciona tão bem ao contrário. Quando muitos íons se acumulam no lado externo do cromatóforo, o anel de proteína ATP sintase vai quebrar ATP em ADP, um processo chamado hidrólise, e os íons fluirão de volta para o lado interno.
"Normalmente, você esperaria muita perda de energia durante este processo, como em qualquer motor feito pelo homem, mas acontece que a ATP sintase tem muito pouco desperdício, "Singharoy disse." Como este motor é projetado para minimizar a perda de energia é a pergunta que começamos a fazer. "
Semelhante a um funileiro desmontando um motor para entender melhor como ele funciona, A equipe de Singharoy quebrou o 300, Enzima de 000 átomos em suas partes constituintes. Com base em décadas de pesquisa em ATP sintase, modelos anteriores, e novos dados experimentais fornecidos por uma equipe japonesa liderada por Takeshi Murata do RIKEN Centre for Life Science Technologies, a equipe construiu e simulou as peças do quebra-cabeça da ATP sintase de forma independente e em conjunto no Titan.
Para capturar processos importantes que ocorrem em escalas de tempo de milissegundos, Singharoy, em colaboração com Christophe Chipot do Centro Nacional Francês de Pesquisa Científica e Mahmoud Moradi da Universidade de Arkansas, implantou o código de dinâmica molecular NAMD estrategicamente. A equipe executou uma estratégia de conjunto, rastreando o movimento de cerca de 1, 000 réplicas de ATP sintase simultaneamente com etapas de 2 femtossegundos, ou 2, 000 trilionésimos de segundo. No total, a equipe acumulou 65 microssegundos (65 milionésimos de segundo) de tempo de simulação, usar essas informações para extrapolar os movimentos que ocorrem ao longo de um milissegundo (1 milésimo de segundo).
Como resultado, a equipe identificou movimentos giratórios anteriormente não documentados no anel de proteína que ajudam a explicar a eficiência do motor molecular. De forma similar, as simulações da equipe capturaram a elasticidade semelhante a um elástico da haste central da enzima. A equipe de Singharoy estimou que, quando emparelhado com o anel de proteína, o talo absorve cerca de 75 por cento da energia liberada durante a hidrólise.
Adicionalmente, simulações do anel de proteína por si só revelaram uma unidade que pode funcionar de forma independente, uma descoberta relatada em experimentos, mas não em detalhes computacionais. "Mesmo na ausência do talo central, o próprio anel de proteína é capaz de hidrólise de ATP. Não é muito eficiente, mas tem a capacidade, "Singharoy disse.
A grande imagem
Depois de simular seu modelo completo de ATP sintase, a equipe da UIUC incorporou a enzima em seu modelo de cromatóforo previamente construído para obter a imagem mais abrangente de um sistema fotossintético até o momento.
Com este painel solar biológico virtual, a equipe poderia medir cada etapa do processo de conversão de energia - da coleta de luz, para transferência de elétron e próton, para a síntese de ATP - e entender melhor seus fundamentos mecânicos.
O cromatóforo da natureza é projetado para baixa intensidade de luz, absorvendo apenas entre 3 e 5 por cento da luz solar em um dia normal. O time, através dos esforços de Sener, descobriram que essa taxa de absorção se traduz em cerca de 300 ATPs por segundo, que é a quantidade de que uma bactéria precisa para se manter viva.
Tendo estudado o design da natureza, a equipe agora queria ver se poderia melhorar. Supondo a mesma quantidade de intensidade de luz, a equipe projetou um cromatóforo artificial com uma composição de proteína decididamente não natural, aumentando a presença de dois tipos de proteínas especializadas. A análise do novo design previu uma triplicação da produção de ATP do sistema fotossintético, abrindo a possibilidade de otimização guiada por humanos do cromatóforo.
"Você poderia modificar geneticamente um cromatóforo ou alterar sua concentração de proteínas, "Singharoy disse." Essas previsões prometem trazer novos desenvolvimentos na fotossíntese artificial. "
De acordo com sua última alocação do INCITE, a equipe da UIUC está se voltando para a conversão de energia em uma forma de vida diferente:os animais. Pegando o que aprendeu com a modelagem da fotossíntese em bactérias roxas, a equipe está modelando a respiração celular, o processo que as células animais usam para converter nutrientes em ATP.
"Você tem pelo menos duas proteínas em comum entre a respiração e a fotossíntese, "disse Singharoy, que continua seu envolvimento com o projeto como professor assistente na Arizona State University. "A questão é quais princípios de design são transportados para os organismos superiores?"
Vida in situ
A simulação do cromatóforo - completo com ATP sintase - marca uma mudança contínua na biofísica computacional a partir da análise de partes individuais de células (por exemplo, proteínas únicas e centenas de átomos) para analisar sistemas celulares inteiros (por exemplo, centenas de proteínas e milhões de átomos).
Schulten, que faleceu em outubro de 2016, entendeu melhor do que a maioria das pessoas a importância de usar computadores para simular a natureza. Em entrevista em 2015, ele expôs seus fundamentos para modelar o cromatóforo. "A motivação é entender uma etapa muito importante da vida na Terra, da qual toda a vida depende hoje. Em termos de energia, 95 por cento da vida na Terra depende da fotossíntese, incluindo humanos, " ele disse.
Schulten também entendeu o marco que uma organela especializada representou no caminho para simular um organismo unicelular completo. "Não temos nada menor do que uma célula que chamaríamos de viva, "disse ele." É a menor entidade viva, e queremos entender isso. "
Com supercomputadores de última geração, incluindo a Cúpula do OLCF, definido para ficar online em 2018, o grupo de pesquisa fundado por Schulten em 1989 está se preparando para assumir o grande desafio de simular uma célula.
Sob a liderança de Tajkhorshid, a equipe planeja simular a primeira célula de um bilhão de átomos, incluindo os componentes básicos de que uma célula precisa para sobreviver e crescer. As melhorias no NAMD e o trabalho que está sendo realizado no programa Center for Accelerated Application Readiness do OLCF estão ajudando a tornar a visão de Schulten e de outros uma realidade.
"Continuamos avançando, "Singharoy disse." Nosso estudo exaustivo de uma organela completa em todos os detalhes de átomos abriu a porta para uma célula completa em todos os detalhes de átomos.