A nanotecnologia permite a visualização de estruturas de RNA em resolução quase atômica
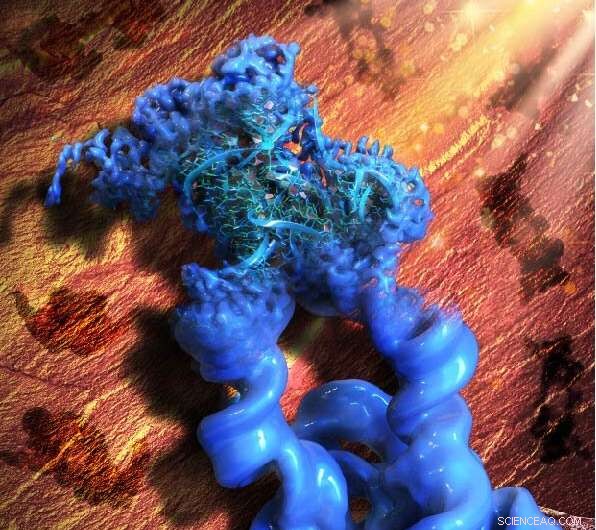
Esta ilustração é inspirada na pintura rupestre paleolítica na caverna de Lascaux, significando a sigla do nosso método, ROCK. Figurativamente, os padrões da arte rupestre no fundo (marrom) são as projeções 2D da construção dimérica projetada do íntron do grupo Tetrahymena I, enquanto o objeto principal na frente (azul) é o mapa 3D crio-EM reconstruído do dímero, com um monômero em foco e refinado à alta resolução que permitiu aos colaboradores construir um modelo atômico do RNA. Crédito:Instituto Wyss da Universidade de Harvard
Vivemos em um mundo feito e dirigido pelo RNA, o irmão igualmente importante da molécula genética DNA. De fato, os biólogos evolucionistas supõem que o RNA existia e se auto-replicava mesmo antes do aparecimento do DNA e das proteínas codificadas por ele. Avanço rápido para os humanos modernos:a ciência revelou que menos de 3% do genoma humano é transcrito em moléculas de RNA mensageiro (mRNA) que, por sua vez, são traduzidas em proteínas. Em contraste, 82% dele é transcrito em moléculas de RNA com outras funções, muitas das quais ainda permanecem enigmáticas.
Para entender o que uma molécula de RNA individual faz, sua estrutura 3D precisa ser decifrada no nível de seus átomos constituintes e ligações moleculares. Os pesquisadores estudaram rotineiramente moléculas de DNA e proteínas, transformando-as em cristais regularmente empacotados que podem ser examinados com um feixe de raios X (cristalografia de raios X) ou ondas de rádio (ressonância magnética nuclear). No entanto, essas técnicas não podem ser aplicadas a moléculas de RNA com quase a mesma eficácia porque sua composição molecular e flexibilidade estrutural as impedem de formar cristais facilmente.
Agora, uma colaboração de pesquisa liderada pelo membro do Wyss Core Faculty Peng Yin, Ph.D. no Wyss Institute for Biologicamente Inspirado Engenharia da Universidade de Harvard, e Maofu Liao, Ph.D. na Harvard Medical School (HMS), relatou uma abordagem fundamentalmente nova para a investigação estrutural de moléculas de RNA. O ROCK, como é chamado, usa uma técnica nanotecnológica de RNA que permite montar várias moléculas de RNA idênticas em uma estrutura altamente organizada, o que reduz significativamente a flexibilidade de moléculas de RNA individuais e multiplica seu peso molecular. Aplicado a RNAs modelo bem conhecidos com diferentes tamanhos e funções como referência, a equipe mostrou que seu método permite a análise estrutural das subunidades de RNA contidas com uma técnica conhecida como microscopia crio-eletrônica (crio-EM). Seu avanço é relatado em
Métodos da Natureza .
“O ROCK está quebrando os limites atuais das investigações estruturais de RNA e permite que estruturas 3D de moléculas de RNA sejam desbloqueadas que são difíceis ou impossíveis de acessar com os métodos existentes e com resolução quase atômica”, disse Yin, que junto com Liao liderou o estudo. . "Esperamos que esse avanço revigore muitas áreas de pesquisa fundamental e desenvolvimento de medicamentos, incluindo o campo florescente da terapia de RNA". Yin também é líder da Molecular Robotics Initiative do Wyss Institute e professor do Departamento de Biologia de Sistemas da HMS.
Ganhando controle sobre o RNA A equipe de Yin no Wyss Institute foi pioneira em várias abordagens que permitem que as moléculas de DNA e RNA se automontem em grandes estruturas com base em diferentes princípios e requisitos, incluindo tijolos de DNA e origami de DNA. Eles levantaram a hipótese de que essas estratégias também poderiam ser usadas para montar moléculas de RNA que ocorrem naturalmente em complexos circulares altamente ordenados, nos quais sua liberdade de flexionar e mover é altamente restrita, ligando-os especificamente. Muitos RNAs se dobram de maneiras complexas, porém previsíveis, com pequenos segmentos emparelhados de bases. O resultado muitas vezes é um "núcleo" estabilizado e "stem-loops" salientes na periferia.
"Em nossa abordagem, instalamos 'kissing loops' que ligam diferentes hastes periféricas pertencentes a duas cópias de um RNA idêntico de uma maneira que permite a formação de um anel estabilizado geral, contendo várias cópias do RNA de interesse", disse Di Liu, Ph.D., um dos dois primeiros autores e bolsista de pós-doutorado no grupo de Yin. "Nós especulamos que esses anéis de ordem superior poderiam ser analisados com alta resolução por crio-EM, que foi aplicado a moléculas de RNA com primeiro sucesso".
Retratando RNA estabilizado No crio-EM, muitas partículas únicas são congeladas em temperaturas criogênicas para evitar movimentos adicionais e, em seguida, visualizadas com um microscópio eletrônico e a ajuda de algoritmos computacionais que comparam os vários aspectos das projeções de superfície 2D de uma partícula e reconstroem sua arquitetura 3D . Peng e Liu se uniram a Liao e seu ex-aluno de pós-graduação François Thélot, Ph.D., o outro co-primeiro autor do estudo. Liao com seu grupo fez contribuições importantes para o rápido avanço do campo crio-EM e a análise experimental e computacional de partículas únicas formadas por proteínas específicas.
"Cryo-EM tem grandes vantagens sobre os métodos tradicionais na visualização de detalhes de alta resolução de moléculas biológicas, incluindo proteínas, DNAs e RNAs, mas o tamanho pequeno e a tendência de movimento da maioria dos RNAs impedem a determinação bem-sucedida de estruturas de RNA. Nosso novo método de montagem de multímeros de RNA resolve esses dois problemas ao mesmo tempo, aumentando o tamanho do RNA e reduzindo seu movimento", disse Liao, que também é Professor Associado de Biologia Celular no HMS. "Nossa abordagem abriu as portas para a determinação rápida da estrutura de muitos RNAs por crio-EM". A integração das abordagens de nanotecnologia de RNA e crio-EM levou a equipe a nomear seu método "crio-EM habilitado para oligomerização de RNA através da instalação de laços de beijo" (ROCK).
Para fornecer prova de princípio para o ROCK, a equipe se concentrou em um grande RNA de íntron de Tetrahymena, um organismo unicelular, e um pequeno RNA de íntron de Azoarcus, uma bactéria fixadora de nitrogênio, bem como o chamado riboswitch FMN . Os RNAs de íntrons são sequências de RNA não codificantes espalhadas pelas sequências de RNAs recém-transcritos e precisam ser "spliced" para que o RNA maduro seja gerado. O riboswitch FMN é encontrado em RNAs bacterianos envolvidos na biossíntese de metabólitos de flavina derivados da vitamina B2. Ao se ligar a um deles, o mononucleotídeo de flavina (FMN), ele muda sua conformação 3D e suprime a síntese de seu RNA mãe.
"A montagem do íntron do grupo Tetrahymena I em uma estrutura semelhante a um anel tornou as amostras mais homogêneas e permitiu o uso de ferramentas computacionais que alavancam a simetria da estrutura montada. Embora nosso conjunto de dados seja relativamente modesto em tamanho, as vantagens inatas do ROCK nos permitiram para resolver a estrutura em uma resolução sem precedentes", disse Thélot. "O núcleo do RNA é resolvido em 2,85 Å [um Ångström é um dez bilhões (US) de um metro e a métrica preferida usada por biólogos estruturais], revelando características detalhadas das bases de nucleotídeos e da espinha dorsal do açúcar. poderia ter chegado lá sem o ROCK - ou pelo menos não sem consideravelmente mais recursos."
O Cryo-EM também é capaz de capturar moléculas em diferentes estados se elas, por exemplo, alterarem sua conformação 3D como parte de sua função. Aplicando ROCK ao RNA do íntron de Azoarcus e ao riboswitch FMN, a equipe conseguiu identificar as diferentes conformações pelas quais o íntron de Azoarcus transita durante seu processo de auto-splicing e revelar a relativa rigidez conformacional do sítio de ligação ao ligante do riboswitch FMN .
"Este estudo de Peng Yin e seus colaboradores mostra elegantemente como a nanotecnologia de RNA pode funcionar como um acelerador para o avanço de outras disciplinas. processos em diferentes tipos de células, tecidos e organismos, e até mesmo permitir novas abordagens de desenvolvimento de medicamentos", disse o diretor fundador da Wyss, Donald Ingber, M.D., Ph.D.
+ Explorar mais Novos insights sobre as estruturas e mecanismos das principais proteínas envolvidas na fotossíntese microbiana
