Visão molecular de um modelo de granulação grossa com base na estrutura original das proteínas de revestimento principal M13. Crédito:SUTD
As simulações atomísticas são uma ferramenta poderosa para estudar o movimento e as interações de átomos e moléculas. Em muitos processos biológicos, efeitos em grande escala, por exemplo, a montagem de grandes vírus em nanopartículas é importante. Os processos de montagem desses grandes vírus são de fundamental importância para o projeto de muitos dispositivos e terapêuticas direcionadas a proteínas virais. Contudo, a escala de tempo e comprimento desses processos de montagem são geralmente muito grandes para simulações em resolução molecular.
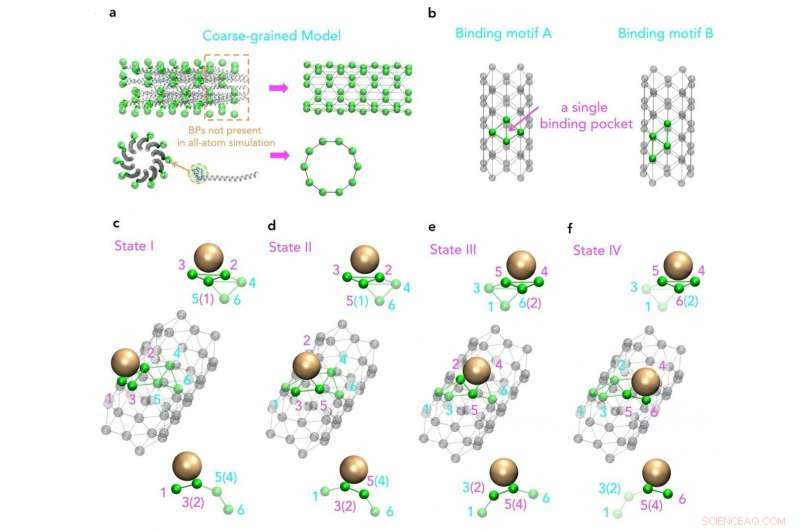
Além disso, embora um aumento no poder de computação permita simulações mais complexas e mais longas, estruturas de vírus, como M13, ainda estão fora de alcance. É por isso que um grupo de pesquisa da Universidade de Tecnologia e Design de Cingapura (SUTD) e do Instituto de Tecnologia de Massachusetts (MIT) desenvolveu um procedimento que vincula processos de montagem em grande escala a simulações moleculares. Professor Assistente Desmond Loke da Ciência do SUTD, O cluster de Matemática e Tecnologia disse:"Para a simulação de M13, começamos com diferentes conjuntos de campos de força. Campos de força adequados foram escolhidos e usados como entradas para simulações de dinâmica de moléculas com o modelo de grão grosso projetado para capturar o padrão-chave do processo de montagem. "
"Embora saibamos que a fabricação baseada em M13 pode ser fundamentalmente impulsionada por interações nanopartícula-peptídeo, que também pode ser um princípio fundamental por trás da bioengenharia do tipo M13, temos pouco conhecimento de como os padrões repetidos de peptídeos terminais curtos em uma superfície M13 estão realmente envolvidos nessas interações. Para estudar isso, idealmente, temos que incluir uma estrutura completa da proteína de revestimento viral, que é uma tarefa difícil para as simulações de dinâmica molecular de última geração, "acrescenta a Dra. Lunna Li, primeiro autor do artigo.
O procedimento permite aos usuários adicionar diferentes tipos de nanopartículas a uma solução, em um nível realista. Inspirado por este procedimento, O professor assistente Loke e seus colegas foram capazes de simular um vírus em grande escala com nanopartículas e dentro de uma solução por cinquenta nanossegundos.
Dr. Li disse, "A estrutura e a solução do vírus contêm cerca de 700, 000 átomos no total. "Considerando a forma e o tamanho das características, a complexidade desta simulação pode ser maior do que qualquer simulação realizada anteriormente.
"Uma simulação realizada em microssegundos teria sido possível se um modelo M13 menor fosse usado, mas pode valer a pena reduzir o tempo para realmente observar como a estrutura completa pode influenciar a montagem entre o M13 e as nanopartículas, "explicou o Assistente Prof Loke.