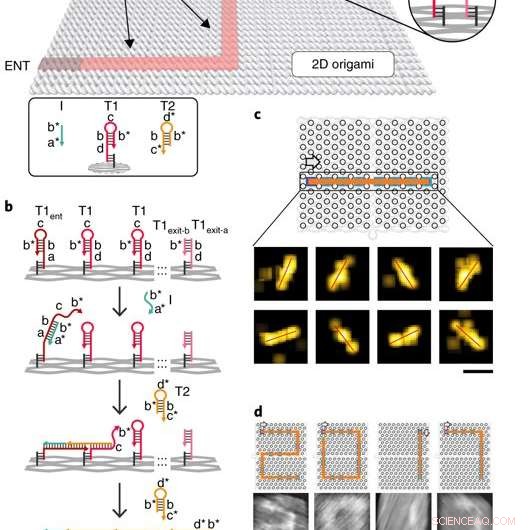
 p Implementando o navegador de DNA de uma única molécula. a) Ilustração esquemática do sistema PSEC. Um gráfico conectado acíclico (ou seja, uma árvore) é construído em um substrato de origami de DNA retangular de 100 x 70 nm ^ 2. T1 e T2 são dois tipos de combustíveis que impulsionam o PSEC na árvore. O iniciador I foi usado para acionar a iniciação do PSEC a partir do vértice de entrada ENT. b) mecanismo de propagação do sistema PSEC. O iniciador I reconhece e abre o grampo de cabelo T1ent no vértice ENT. O grampo aberto então captura e abre um grampo T2 do ambiente para iniciar a cascata. c) Uma linha reta pavimentada por PSEC e visualizada com DNA-PAINT. d) Quatro dígitos pavimentados de imagens de "2017" usando microscopia de força atômica (AFM). Os grampos T1 nos pontos dos cantos foram modificados para evitar extensões indesejadas. As setas indicam a direção de propagação. Crédito: Materiais da Natureza , doi:10.1038 / s41563-018-0205-3.
p Implementando o navegador de DNA de uma única molécula. a) Ilustração esquemática do sistema PSEC. Um gráfico conectado acíclico (ou seja, uma árvore) é construído em um substrato de origami de DNA retangular de 100 x 70 nm ^ 2. T1 e T2 são dois tipos de combustíveis que impulsionam o PSEC na árvore. O iniciador I foi usado para acionar a iniciação do PSEC a partir do vértice de entrada ENT. b) mecanismo de propagação do sistema PSEC. O iniciador I reconhece e abre o grampo de cabelo T1ent no vértice ENT. O grampo aberto então captura e abre um grampo T2 do ambiente para iniciar a cascata. c) Uma linha reta pavimentada por PSEC e visualizada com DNA-PAINT. d) Quatro dígitos pavimentados de imagens de "2017" usando microscopia de força atômica (AFM). Os grampos T1 nos pontos dos cantos foram modificados para evitar extensões indesejadas. As setas indicam a direção de propagação. Crédito: Materiais da Natureza , doi:10.1038 / s41563-018-0205-3.
p O campo da nanorrobótica inteligente é baseado na grande promessa de dispositivos moleculares com capacidade de processamento de informações. Em um novo estudo que apóia a tendência de portadores de informações baseados em DNA, cientistas desenvolveram um sistema de navegação de DNA que pode realizar uma única molécula, paralelo, operações de pesquisa em profundidade em uma plataforma de origami bidimensional. p As operações de Pathfinding com navegadores de DNA usam um processo de cascata de troca de fita localizada, iniciado em um local de gatilho exclusivo na plataforma de origami. A progressão automática ao longo dos caminhos é ativada por grampos de DNA contendo uma sequência de travessia universal. Por design, cada navegador de uma única molécula pode explorar de forma autônoma qualquer um dos caminhos possíveis por meio de uma árvore enraizada de 10 vértices construída no estudo. Os labirintos eram equivalentes a uma árvore com entrada pela raiz e saída por uma das folhas. O estudo conduzido por Jie Chao e colegas de trabalho resultou na exploração de todos os caminhos percorridos pelos navegadores de DNA para extrair um caminho de solução específico que conectasse um determinado par de vértices inicial e final no labirinto. Como resultado, o caminho da solução foi traçado claramente na plataforma de origami e ilustrado usando imagens de uma única molécula. A abordagem agora está publicada em
Materiais da Natureza , detalhando a realização de materiais moleculares com funções computacionais biomoleculares incorporadas para operar no nível da única molécula com potencial para projetar nanorrobôs inteligentes para futuras aplicações na indústria e na medicina.
p Ferramentas moleculares sofisticadas foram usadas no passado para criar máquinas moleculares que convertem produtos químicos, energia fotônica ou elétrica em movimentos rotativos ou lineares em nanoescala. Por exemplo, O movimento browniano em nanoescala pode ser convertido de forma controlada em movimentos direcionados em nanomáquinas baseadas em DNA usando reações de hibridização de DNA. Essas máquinas baseadas em DNA operam de forma autônoma, seguindo um 'programa molecular' embutido pré-projetado como uma reação em cascata acionada manualmente por meio de um estímulo externo para cada etapa da operação.
p O foco do campo mudou progressivamente para atualizar circuitos lógicos baseados em DNA usando aptâmeros e DNAzymes para projetar portas lógicas moleculares. Por exemplo, em 2006, Stojanovic e colegas de trabalho integraram mais de 100 portas lógicas de DNA para criar uma automação chamada MAYA-II para jogar Tic-Tac-Toe. Estudos anteriores demonstraram um sistema de computação livre de enzimas baseado em reações em cadeia de hibridização (HCR) para criar portas lógicas e circuitos lógicos para um desempenho mais robusto e eficiente do que os sistemas originais.
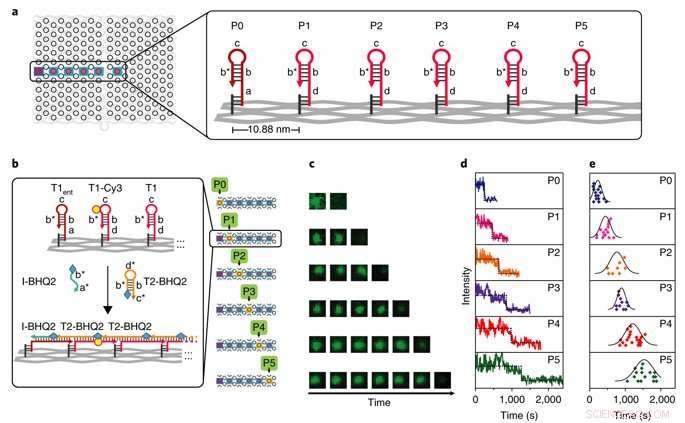 p Caracterização de uma única molécula da cinética PSEC. a) Uma linha reta no meio do origami de DNA foi usada como base de teste. O vértice P0 é o ponto de partida e os vértices P1-P5 são pontos intermediários. b) Detalhes do projeto de seis testes paralelos para medir a cinética com TIRF resolvido no tempo. T2 marcado com BHQ2 foi usado para extinguir T1 marcado com Cy3, montagem de um perfil cinético em cada etapa. O exemplo ilustrado mostra o mecanismo de têmpera. c) As imagens TIRF de exemplo mostram a fluorescência mudando com o tempo nos seis testes paralelos de P0 a P5. d) Traços de fluorescência de molécula única típicos usados para monitorar eventos de extinção que ocorrem nos vértices P0 a P5. e) Gráficos de dispersão mostrando análise estatística da distribuição de comprimento por caminho. Crédito: Materiais da Natureza , doi:10.1038 / s41563-018-0205-3.
p Caracterização de uma única molécula da cinética PSEC. a) Uma linha reta no meio do origami de DNA foi usada como base de teste. O vértice P0 é o ponto de partida e os vértices P1-P5 são pontos intermediários. b) Detalhes do projeto de seis testes paralelos para medir a cinética com TIRF resolvido no tempo. T2 marcado com BHQ2 foi usado para extinguir T1 marcado com Cy3, montagem de um perfil cinético em cada etapa. O exemplo ilustrado mostra o mecanismo de têmpera. c) As imagens TIRF de exemplo mostram a fluorescência mudando com o tempo nos seis testes paralelos de P0 a P5. d) Traços de fluorescência de molécula única típicos usados para monitorar eventos de extinção que ocorrem nos vértices P0 a P5. e) Gráficos de dispersão mostrando análise estatística da distribuição de comprimento por caminho. Crédito: Materiais da Natureza , doi:10.1038 / s41563-018-0205-3.
p No presente estudo de Chao et al, o mesmo princípio básico do esquema de reação HCR foi usado em um contexto computacional diferente para desenvolver um sistema navegador de DNA de uma única molécula. A plataforma explorou todos os caminhos possíveis através de um gráfico de árvore projetado em uma estrutura de origami como um labirinto simplesmente conectado sem caminhos cíclicos. Essas estruturas de origami de DNA são nanoestruturas portadoras de informações por natureza, com geometria em nanoescala bem definida. O labirinto pode ser explorado por cascata de troca de fita proximal (PSEC) com base em reações em cadeia de hibridização. Os pesquisadores demonstraram que um sistema com um grande número de navegadores de DNA de molécula única poderia conduzir coletivamente a pesquisa em profundidade paralela (PDFS) na árvore para realizar a resolução de labirintos com eficiência em origami 2-D. Inicialmente, os pesquisadores conduziram estudos para testar o projeto PSEC.
p O sistema de cascata de troca de fita proximal (PSEC) (princípio de funcionamento do navegador de DNA) foi facilitado em um substrato de origami retangular feito de três componentes, que incluiu a implementação física de um gráfico de árvore, fios completos e um fio iniciador. As áreas vazias sem extensões básicas correspondiam às paredes do labirinto, prevenir a propagação da cascata de troca de fita. The entrance and exit were defined and denoted as ENT and EXIT respectively. In the second component, two types of DNA hairpins, T1 and T2, were used as fuels to drive the PSEC on the tree graph. The two hairpins coexisted metastably in solution to hybridize and fuel the PSEC process with free energy .
p Por design, information only propagated through the network in the presence of an initiator (Initiator I). Upon addition of initiator I, PSEC was conducted and observed using atomic force microscopy (AFM). To visualize an established formation, the scientists enabled DNA-navigator-based formation of the number 2017 as a proof-of-principle. Another technique known as DNA-PAINT was employed as a single-molecule, super-resolution imaging technique to reveal molecular features at the nanoscale to further substantiate the PSEC-based path paving process. The on-origami PSEC was highly specific, without intra- or inter-origami crosstalk.
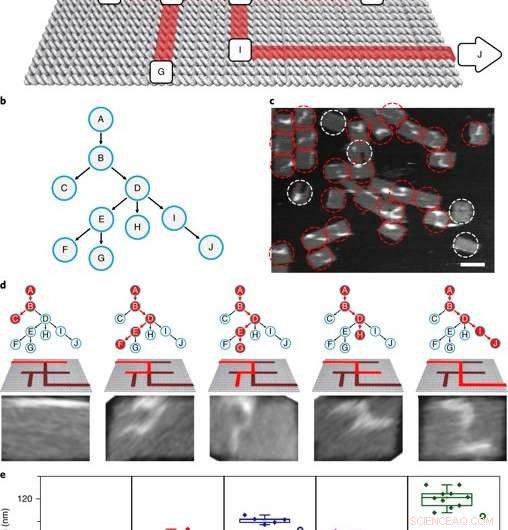 p The PSEC-driven graph traversal on a maze. a) A maze design with 10 vertices. Arrows indicate the entrance vertex A and exit vertex J. b) The maze is equivalent to a rooted tree with 10 vertices. The entrance vertex A corresponds to the root of the tree. c) An AFM image showed the result of a transversal experiment generating all possible paths. In this DNA computing implementation of a PDFS algorithm, a vast number of PSEC events simultaneously occurred to realize the graph traversal on the maze. PSEC ending at the exit or deadends were highlighted in red circles. Invalid structures were highlighted with white circles. d) Typical paths found in the mixture seen from left to right. Only P ABDIJ was the correct solution to the maze. e) Scatter plots showing the statistical analysis of the length distribution for each path. Crédito: Materiais da Natureza , doi:10.1038/s41563-018-0205-3.
p The PSEC-driven graph traversal on a maze. a) A maze design with 10 vertices. Arrows indicate the entrance vertex A and exit vertex J. b) The maze is equivalent to a rooted tree with 10 vertices. The entrance vertex A corresponds to the root of the tree. c) An AFM image showed the result of a transversal experiment generating all possible paths. In this DNA computing implementation of a PDFS algorithm, a vast number of PSEC events simultaneously occurred to realize the graph traversal on the maze. PSEC ending at the exit or deadends were highlighted in red circles. Invalid structures were highlighted with white circles. d) Typical paths found in the mixture seen from left to right. Only P ABDIJ was the correct solution to the maze. e) Scatter plots showing the statistical analysis of the length distribution for each path. Crédito: Materiais da Natureza , doi:10.1038/s41563-018-0205-3.
p Kinetics of the process were investigated at the single-molecule level in the study using time-resolved total internal reflection fluorescence microscopy (TIRF) in a setup with a prescribed starting point (P0) and five intermediate steps (P1-P5). Fluorescence in the setup was quenched using fluorescence resonance energy transfer (FRET), and the cascade was observed in real time by recording fluorescence signals continuously. The average speed of propagation was recorded to be 2.46 nm per minute, propagation across the straight line (54.4 nm) took approximately 22 minutes.
p The scientists then constructed the main model maze with 10 vertices that included an entrance vertex A and an exit vertex J, three junctions (B, D, E) an intermediate vertex (I) and four dead ends (C, F, G, H). Each path of the maze that was equivalent to a tree with 10 vertices was investigated using DNA investigators starting at root A. The PSEC reactions produced a mixture of various paths on the maze, confirmed with AFM. Each individual PSEC could progress on one of the five possible paths. Statistical analysis of the length distribution showed that the measured paths coincided well with the values predicted.
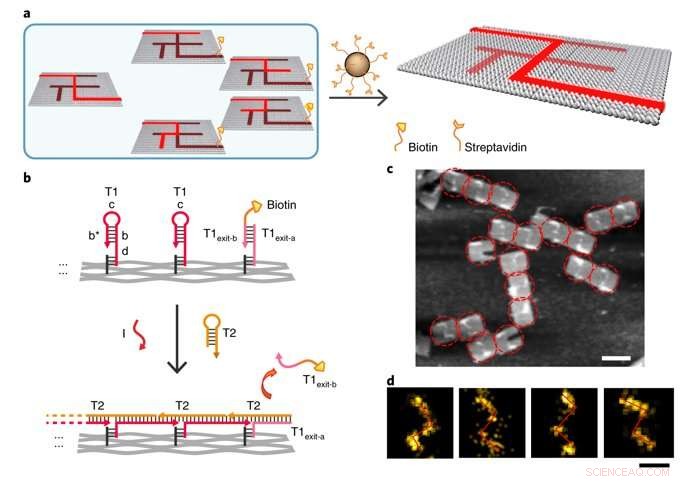 p Single-molecule DNA navigators for maze-solving. a) Schematic illustration of magnetic bead-based selection. Exit vertex J is labelled with biotin to enable differentiation between the correct and wrong paths. Only if the PSEC reaches the correct exit J, would the biotin-modified T1exit-B strand be released. All wrong paths could be captured and removed by the Streptavidin-modified magmatic beads therefore. b) Details of the release of biotin modification at the exit J by the PSEC. c) AFM characterization of the correct solution after selection. The remaining structures all showed the correct solution path PABDIJ. d) Single-molecule and class-averaged DNA-PAINT characterization of the correct solution after selection. Crédito: Materiais da Natureza , doi:10.1038/s41563-018-0205-3.
p Single-molecule DNA navigators for maze-solving. a) Schematic illustration of magnetic bead-based selection. Exit vertex J is labelled with biotin to enable differentiation between the correct and wrong paths. Only if the PSEC reaches the correct exit J, would the biotin-modified T1exit-B strand be released. All wrong paths could be captured and removed by the Streptavidin-modified magmatic beads therefore. b) Details of the release of biotin modification at the exit J by the PSEC. c) AFM characterization of the correct solution after selection. The remaining structures all showed the correct solution path PABDIJ. d) Single-molecule and class-averaged DNA-PAINT characterization of the correct solution after selection. Crédito: Materiais da Natureza , doi:10.1038/s41563-018-0205-3.
p To prevent the navigators from propagating through a wrong path with dead ends, the scientists designed a streptavidin-biotin tag-based method to selectively eliminate inaccurate path navigation. Only the correct path (P
ABDIJ ) was followed in the maze therefore. The computational context used in the study allowed exploration of paths through tree graphs defined on the origami. The autonomous path explored by the DNA navigators proceeded unidirectionally and irreversibly, turning at junctions and corners on the origami platform as they were designed to. The design enabled parallel depth first search (PDFS) allowing each DNA navigator to individually explore any one of the paths through the given graph at a defined speed, greater than that previously achieved.
p The main advantage of the described biomolecular computer schemes in comparison to conventional electronic computing is they can be interfaced directly with biologically relevant processes. Como resultado, the scientists envision translational biomedical sensing and decision-making platforms with DNA origami and single-molecule diagnostics using decision trees. Future applications will also include simple sensors or those coupled to a molecular actuator to trigger downstream molecular cascades. p © 2018 Science X Network



