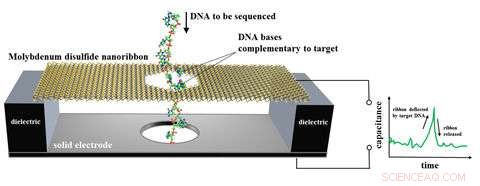
O projeto proposto pelo NIST para um sequenciador de DNA baseado em um sensor eletrônico de movimento. Uma fita em nanoescala de dissulfeto de molibdênio é suspensa sobre um eletrodo de metal e imersa em água. DNA de fita simples, contendo uma cadeia de bases (bits de código genético), é enfiado através de um orifício na fita, que flexiona apenas quando uma base de DNA emparelha com e, em seguida, se separa de uma base complementar afixada no orifício. O movimento da membrana é detectado como um sinal elétrico. Simulações numéricas e estimativas teóricas mostram que a membrana seria de 79 a 86 por cento precisa na identificação de bases de DNA em uma única medição a velocidades de até 70 milhões de bases por segundo. Crédito:NIST
Pesquisadores do Instituto Nacional de Padrões e Tecnologia (NIST) e colaboradores propuseram um projeto para o primeiro sequenciador de DNA baseado em um nanosensor eletrônico que pode detectar movimentos minúsculos tão pequenos quanto um único átomo.
O dispositivo proposto - um tipo de capacitor, que armazena carga elétrica - é uma pequena fita de dissulfeto de molibdênio suspensa sobre um eletrodo de metal e imersa em água. A fita tem 15,5 nanômetros (nm, bilionésimos de metro) de comprimento e 4,5 nm de largura. DNA de fita simples, contendo uma cadeia de bases (bits de código genético), é passado por um orifício de 2,5 nm de largura na fita fina. A fita flexiona apenas quando uma base de DNA emparelha com e, em seguida, se separa de uma base complementar afixada no orifício. O movimento da membrana é detectado como um sinal elétrico.
Conforme descrito em um novo artigo, a equipe do NIST fez simulações numéricas e estimativas teóricas para mostrar que a membrana teria 79 a 86 por cento de precisão na identificação de bases de DNA em uma única medição a velocidades de até 70 milhões de bases por segundo. Os circuitos integrados detectariam e mediriam os sinais elétricos e identificariam as bases. Os resultados sugerem que tal dispositivo pode ser rápido, sequenciador de DNA preciso e econômico, de acordo com o jornal.
Sequenciamento convencional, desenvolvido na década de 1970, envolve separar, copiando, rotulando e remontando pedaços de DNA para ler a informação genética. Os métodos mais novos incluem o sequenciamento automatizado de muitos fragmentos de DNA de uma só vez - ainda caro - e novos conceitos de "sequenciamento de nanoporos". Por exemplo, o mesmo grupo do NIST recentemente demonstrou a ideia de sequenciar DNA passando-o por um nanoporo de grafeno, e medir como as propriedades eletrônicas do grafeno respondem à tensão.
A última proposta do NIST se baseia em uma fina película de dissulfeto de molibdênio - um estável, material em camadas que conduz eletricidade e costuma ser usado como lubrificante. Entre outras vantagens, este material não adere ao DNA, o que pode ser um problema com o grafeno. A equipe do NIST sugere que o método pode até funcionar sem um nanoporo - um design mais simples - ao passar o DNA pela borda da membrana.
"Esta abordagem potencialmente resolve o problema com o DNA grudado no grafeno se inserido incorretamente, porque esta abordagem não usa grafeno, período, "O teórico do NIST e autor principal Alex Smolyanitsky disse." Outra grande diferença é que, em vez de confiar nas propriedades do grafeno ou de qualquer material particular usado, lemos os movimentos eletricamente de uma maneira mais fácil, formando um capacitor. Isso torna qualquer membrana condutora de eletricidade adequada para a aplicação. "
Boris Yakobson, especialista em nanomateriais, da Rice University, um co-autor no artigo, sugeriu a ideia do capacitor. O suporte computacional foi fornecido pela Universidade de Groningen, na Holanda.
O DNA tem quatro bases. Para as simulações, citosina (C), que naturalmente se emparelha com guanina (G), está ligado ao interior do poro. Quando um pedaço de DNA passa pelo poro, qualquer G na fita temporariamente se anexa ao C incorporado, puxando a nanofita e sinalizando o eletrodo. A sequência de DNA é determinada medindo como e quando blips elétricos variam ao longo do tempo. Para detectar todas as quatro bases, quatro nanofitas, cada um com uma base diferente ligada ao poro, pode ser empilhado verticalmente para criar um sensor de DNA integrado.
A fita de dissulfeto de molibdênio é flexível o suficiente para se deformar de forma mensurável em resposta às forças necessárias para quebrar um par de DNA, mas rígido o suficiente para ter menos continuidade, movimento sem sentido do que o grafeno, reduzindo potencialmente o ruído indesejado nos sinais de sequenciamento. A deflexão da fita é extremamente pequena, na ordem de um angstrom, o tamanho de um átomo de hidrogênio. Sua força de tração é da ordem de 50 piconewtons, ou trilionésimos de newton, o suficiente para quebrar as delicadas ligações químicas entre as bases do DNA.
Os pesquisadores estimaram como o dispositivo funcionaria em um circuito integrado e descobriram que as correntes de pico através do capacitor eram mensuráveis (50 a 70 picoamperes), mesmo para as pequenas nanofitas estudadas. Espera-se que os picos atuais sejam ainda maiores em sistemas físicos. O tamanho do dispositivo pode ser ajustado para tornar ainda mais fácil medir os sinais de sequenciamento.
Os autores do NIST esperam construir uma versão física do dispositivo no futuro. Para aplicações práticas, a tecnologia microfluídica de sequenciamento de DNA do tamanho de um chip pode ser combinada com a eletrônica em um único dispositivo pequeno o suficiente para ser portátil.