Puxe com cuidado:uma fita de DNA deve ser conduzida suavemente através de um nanoporo
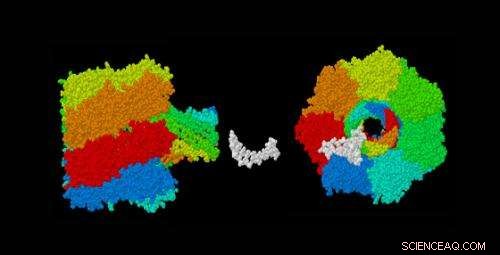 p Esta é uma imagem do poro de alfa-hemolisina (composto por 7 subunidades idênticas em 7 cores) e DNA de fita simples 12-mer (em branco) na mesma escala para ilustrar os efeitos do DNA na condutância ao se mover através de um nanoporo. Abaixo está uma visão ortogonal das mesmas moléculas. Imagem criada em 5 de outubro de 2008 por George Church usando Rasmol_2.6 e coordenadas de 7AHL.pdb e 1BNA.pdb Crédito:George Church
p Esta é uma imagem do poro de alfa-hemolisina (composto por 7 subunidades idênticas em 7 cores) e DNA de fita simples 12-mer (em branco) na mesma escala para ilustrar os efeitos do DNA na condutância ao se mover através de um nanoporo. Abaixo está uma visão ortogonal das mesmas moléculas. Imagem criada em 5 de outubro de 2008 por George Church usando Rasmol_2.6 e coordenadas de 7AHL.pdb e 1BNA.pdb Crédito:George Church
p Não é fácil conduzir longas cadeias de moléculas - como o DNA - através de um "nanoporo" (um poro que tem apenas alguns milionésimos de milímetro de largura) porque elas tendem a se emaranhar. Uma simulação realizada por um grupo internacional de cientistas - entre os quais pesquisadores do SISSA - sugeriu uma solução:é melhor "puxar" suavemente sem aplicar muita força, caso contrário, a molécula pode ser interrompida devido ao atrito excessivo. Essa é uma observação importante para criar métodos inovadores de sequenciamento de DNA. p Conforme a nanotecnologia avança, torna-se cada vez mais importante conhecer em detalhes a dinâmica do nanomundo (o mundo na escala de um milionésimo de milímetro). O que acontece, por exemplo, quando tentamos conduzir um polieletrólito (uma longa cadeia de moléculas eletricamente carregadas, como o DNA) através de um nanoporo se os nós causarem o travamento do processo de translocação? Não é uma pergunta sem sentido, porque agora um novo método de sequenciamento de DNA para analisar eletroquimicamente cada fita, conduzindo-a através de um nanopore, está sendo desenvolvido. Uma vez que esses fios tendem a se emaranhar se forem muito longos, Angelo Rosa da International School for Advanced Studies e seus colegas decidiram estudar a dinâmica dessa translocação teoricamente, realizando uma simulação.
p O modelo escolhido pelos cientistas mostrou que o travamento não é causado pela mera presença do nó, mas pela relação entre o atrito e a força aplicada para conduzir a molécula para a lacuna. "O resultado não é tão óbvio se comparado ao que acontece em um nível macro, "explicou Cristian Micheletti, pesquisador do SISSA e um dos autores do artigo publicado em
Cartas de revisão física . "Os nós introduzem um atrito efetivo que aumenta com a força aplicada e puxa o polímero para o outro lado do nanoporo. A translocação só é interrompida acima de uma força limite".
p “De acordo com o que observamos na simulação, para evitar a obstrução do poro e a interrupção da translocação, a força aplicada deve ser controlada, sem puxar muito ", explicou Rosa.
p Este estudo é apenas o primeiro passo. Para obter detalhes quantitativos sobre esse processo (o que é esse limite e como a força deve ser medida para maximizar a eficácia desse método de sequenciamento), exames mais aprofundados serão necessários tanto a nível teórico (o modelo desenvolvido por Rosa, Di Ventra e Micheletti é mesoscópico, não atomístico) e ao nível experimental.
p
Mais em detalhes ...
p Nanoporesequenciamento é uma técnica inovadora, uma alternativa aos métodos mais tradicionais, como PCA. Este método envolve separar as duas fitas de nucleobases que constituem a dupla hélice do DNA e analisá-las uma a uma. Cada fita é conduzida através de um nanoporo conforme as variações elétricas na translocação são registradas. Esse é um método eletroquímico:alterações no campo elétrico dão informações sobre a composição química da molécula que passa pelo poro e a composição é então reconstruída. Até agora, este método produziu bons resultados com fragmentos curtos de DNA, enquanto as dificuldades foram encontradas para os mais longos, por causa dos nós. É por isso que estudos como Rosa, Di Ventra e Micheletti são um passo importante para aumentar sua eficiência.
