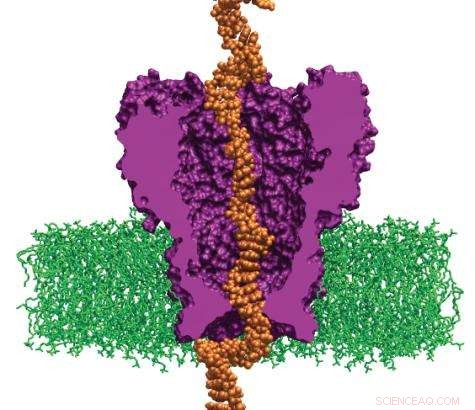
 p Os cientistas simulam a interação do DNA com uma proteína projetada. O sistema pode desacelerar as fitas de DNA que viajam através dos poros o suficiente para ler o genoma individual de um paciente. (Imagem cortesia de Aleksei Aksimentiev)
p Os cientistas simulam a interação do DNA com uma proteína projetada. O sistema pode desacelerar as fitas de DNA que viajam através dos poros o suficiente para ler o genoma individual de um paciente. (Imagem cortesia de Aleksei Aksimentiev)
p (PhysOrg.com) - O Projeto Genoma Humano pavimentou o caminho para a genômica, o estudo do genoma de um organismo. A genômica personalizada pode estabelecer a relação entre as variações da sequência de DNA entre os indivíduos e suas condições de saúde e respostas a medicamentos e tratamentos. Para tornar o sequenciamento do genoma um procedimento de rotina, Contudo, o tempo deve ser reduzido para menos de um dia e o custo para menos de $ 1, 000 - um feito que não é possível com o conhecimento e as tecnologias atuais. p Em 2008, uma equipe de pesquisa liderada por Aleksei Aksimentiev, professor assistente no departamento de física da Universidade de Illinois-Urbana-Champaign, iniciou um projeto para criar máquinas de sequenciamento do genoma pessoal que serão mais acessíveis aos hospitais. Usando o Jaguar do Oak Ridge National Laboratory, um dos supercomputadores mais rápidos do mundo, Aksimentiev e sua equipe estão desenvolvendo uma abordagem nanopore, que promete uma redução drástica de tempo e custos para o sequenciamento de DNA. A pesquisa revela a forma do DNA movendo-se através de um único nanopore - um poro de proteína com um bilionésimo de metro de largura que atravessa uma membrana. Conforme o DNA passa pelo poro, a sequência de nucleotídeos (blocos de construção do DNA) é lida por um detector.
p “O principal obstáculo do sequenciamento usando as gerações mais antigas de nanoporos biológicos e sintéticos era a incapacidade de identificar a sequência de DNA para resolução de nucleotídeo único, "Aksimentiev disse." Os nucleotídeos passaram muito rapidamente através do nanopore para os cientistas sequenciarem o DNA. "
p O grupo de Aksimentiev usa o nanopore MspA, uma proteína projetada. Sua sequência deve ser alterada para se ligar mais fortemente à fita de DNA em movimento. MspA é uma plataforma ideal para o sequenciamento de DNA porque os cientistas agora podem medir barragens nos poros, o que poderia retardar a jornada do DNA através da proteína. Alterar a proteína MspA para otimizar represas é demorado e caro em um laboratório, mas simples em um computador. Por exemplo, para alterar a proteína de qualquer forma, os cientistas devem determinar se a mutação específica que introduzem é estável e se a ideia é razoável. Portanto, os cientistas primeiro simulam o MspA para decidir sobre uma mutação para induzir e testar ideias de alto risco antes de implementá-las em um experimento.
p A equipe de pesquisa usa o código NAMD, que calcula os estados mínimos de energia dos átomos em um grande sistema biomolecular e é um indicador de quais formatos as moléculas seriam mais confortáveis em assumir. A equipe primeiro constrói um modelo da proteína MspA submersa em uma bicamada lipídica e solução eletrolítica. Uma fita de DNA de uma sequência de nucleotídeos desejada é então passada através do nanoporo MspA. Em seguida, os cientistas simulam o efeito de um campo elétrico conduzindo íons e DNA através do nanopore MspA.
p A simulação emprega dinâmica molecular, ou cálculos do movimento de cada átomo em um sistema molecular seguindo as leis físicas da natureza, para imitar o sistema experimental. Os resultados das simulações podem ser comparados diretamente aos de experimentos porque ambas as abordagens medem a corrente iônica, de acordo com Aksimentiev. Conhecendo as posições de cada átomo e íon de DNA, os cientistas ganham uma vantagem - eles podem otimizar o sequenciamento de nanoporos usando um design racional para produzir um poro que abraça o DNA com mais força, retardando a jornada da molécula através do poro a uma velocidade que permite a resolução de um único nucleotídeo.
p O trabalho de sequenciamento é financiado pelo National Human Genome Research Institute dos National Institutes of Health. O desenvolvimento do método do projeto é financiado em parte pela National Science Foundation. Os colaboradores do projeto incluem dois grupos experimentais:um liderado por Jens Gundlach na University of Washington-Seattle e o outro por Michael Niederweis na University of Alabama-Birmingham.
p A pesquisa recebeu 10 milhões de horas de processador no Jaguar por meio do Impacto Computacional Inovador e Novo na Teoria e Experimento, ou INCITE, programa, que concede alocações consideráveis em alguns dos supercomputadores mais poderosos do mundo para projetos que abordam grandes desafios em ciência e engenharia. Com a alocação do INCITE, os cientistas foram capazes de reproduzir as represas no nanoporo de MspA para o tipo de nucleotídeos de DNA confinados a ele, retardando o movimento da sequência através do nanopore.
p "Fizemos um estudo piloto em várias variantes do nanoporo MspA e observamos uma redução considerável da velocidade da fita de DNA, "Aksimentiev disse." Estes resultados muito preliminares sugerem que alcançar uma redução de 100 vezes da velocidade do DNA, que deve ser suficiente para ler a sequência de DNA com resolução de nucleotídeo único, está ao seu alcance. Estudos futuros serão direcionados para esse objetivo. "
p A equipe espera atingir o objetivo deste projeto até 2013 e planeja realizar uma série de projetos spin-off interessantes, Aksimentiev disse. A capacidade de tornar o sequenciamento do genoma acessível permitirá programas como o Projeto Genoma do Câncer, que caracteriza as mutações de DNA em células cancerosas em vários tecidos ao longo de todos os estágios de desenvolvimento do câncer.
