Sondando interações entre moléculas pequenas e RNA observando a FLORESTA
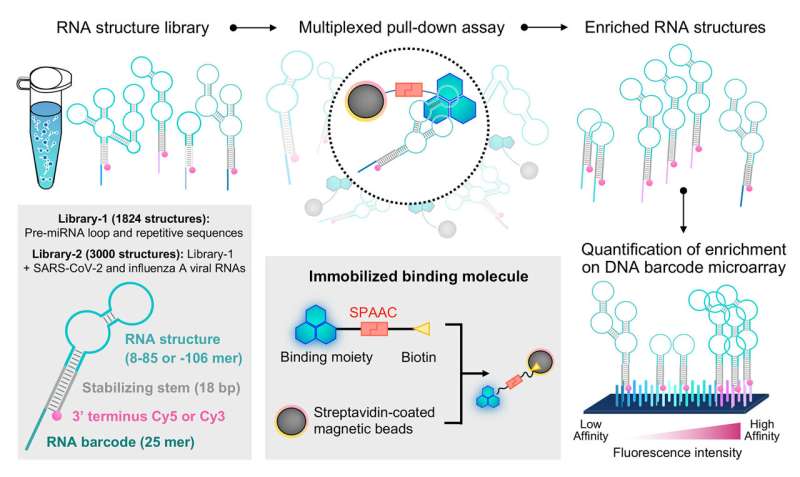 Crédito:Química das Comunicações (2024). DOI:10.1038/s42004-024-01181-8
Crédito:Química das Comunicações (2024). DOI:10.1038/s42004-024-01181-8
Uma equipe de pesquisadores demonstrou recentemente a utilidade de empregar um sistema de triagem previamente estabelecido para sondar as interações entre pequenas moléculas e RNA. O estudo deles foi publicado em Communications Chemistry .
