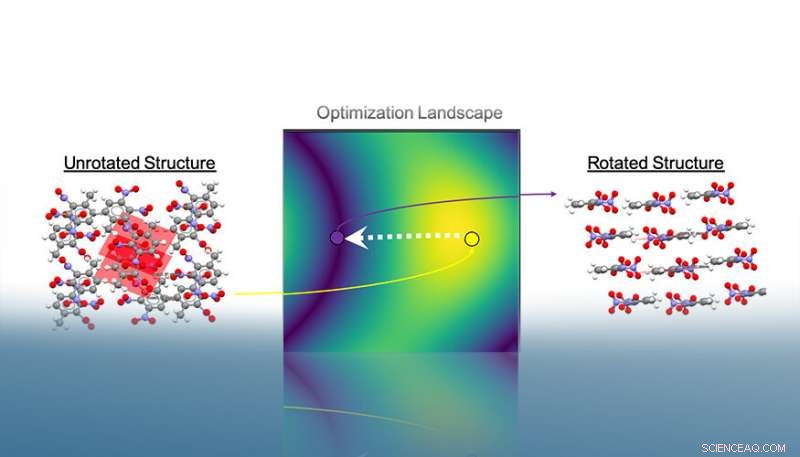
Autopack gira estruturas de cristal no espaço 3D para minimizar a área projetada de suas moléculas. Após a convergência, é possível extrair o motivo de empacotamento associado ao cristal com base em ângulos interplanares relativos. Neste exemplo, as pilhas encontradas após o procedimento de otimização indicam o motivo de empacotamento beta da estrutura. Crédito:Laboratório Nacional Lawrence Livermore
Quer os químicos orgânicos estejam trabalhando no desenvolvimento de novas energias moleculares ou criando novos medicamentos de sucesso na indústria farmacêutica, cada um está procurando como otimizar a estrutura química de uma molécula para atingir as propriedades desejadas.
Parte dessa otimização inclui o motivo de empacotamento de um cristal molecular, um padrão percebido em como as moléculas se orientam em relação umas às outras dentro de uma estrutura cristalina. Os conjuntos de dados atuais de motivos de embalagem permaneceram pequenos por causa dos processos de etiquetagem manual intensivos e esquemas de etiquetagem insuficientes.
Para ajudar a resolver este problema, uma equipe de materiais do Lawrence Livermore National Laboratory (LLNL) e cientistas da computação desenvolveram um pacote disponível gratuitamente, Autopack, que formaliza o processo de rotulagem de motivos de embalagem e pode processar e rotular automaticamente os motivos de embalagem de milhares de estruturas cristalinas moleculares. A pesquisa aparece no Journal of Chemical Information and Modeling .
Estudos de engenharia de cristal em pequena escala nos últimos 30 anos sugerem que, embora a previsão de estruturas cristalinas experimentais a partir de uma estrutura química sozinha permaneça fora de alcance, pode haver relações entre as estruturas químicas das moléculas e um atributo específico da estrutura cristalina que elas adotam, denominado motivo de empacotamento.
O motivo de empacotamento de um cristal molecular é um conceito importante para aplicações de energia e eletrônica orgânica devido às correlações observadas entre os motivos de empacotamento dos cristais moleculares e propriedades de desempenho de interesse, que incluem insensibilidade para explosivos moleculares e transporte de carga para semicondutores moleculares.
Nenhum método formalizado e de código aberto de atribuição de motivos de embalagem foi criado até agora. Em vez de, motivos de empacotamento são atribuídos a cristais moleculares simplesmente pela avaliação humana de uma estrutura cristalina e julgamento, resultando em conjuntos de dados pequenos e barulhentos.
"Na era do aprendizado de máquina, a capacidade de criar grandes, conjuntos de dados rotulados de motivos de empacotamento de cristal molecular são agora especialmente importantes, "disse o cientista de dados do LLNL Donald Loveland, autor principal do artigo. "Esses esforços podem gerar modelos que podem prever os motivos de empacotamento apenas a partir da estrutura química das moléculas, o que ajudaria os químicos orgânicos a priorizar as sínteses de novas moléculas com base nas propriedades e motivos de empacotamento desejados. "
O novo trabalho do LLNL usa um algoritmo de otimização eficiente que contorna muitos problemas encontrados em métodos de rotulagem de motivos de embalagem propostos anteriormente, levando a novos resultados de última geração quando testados em um conjunto de dados com curadoria de LLNL.
Por meio do Autopack, pesquisadores conseguiram gerar um conjunto de dados de quase 10, 000 motivos de embalagem para um conjunto de moléculas energéticas e semelhantes a energia de interesse para o laboratório, uma tarefa que antes teria sido impossível. Para contexto, a literatura anterior permaneceu limitada à ordem de 100 moléculas devido à natureza tediosa e demorada da marcação manual. A análise inicial deste novo conjunto de dados sugere tendências complexas entre as interações intermoleculares, Conformações moleculares 3-D e motivos de empacotamento adotados atualmente inexplorados no campo, fornecendo orientação sobre as próximas etapas para pipelines de engenharia de cristal.
O código está disponível gratuitamente no Escritório de Inovações e Parcerias do Laboratório.