Modelos de proteínas de resolução atômica revelam novos detalhes sobre a ligação de proteínas
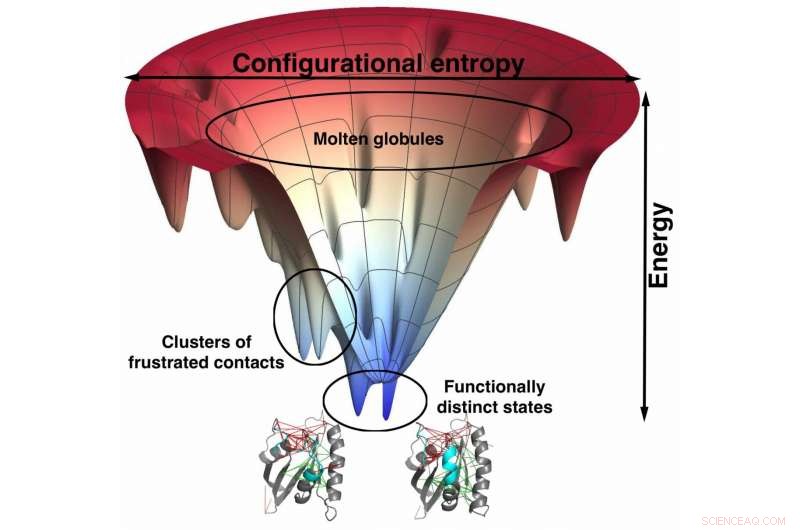 p Modelos em escala atômica por cientistas da Rice University baseados naqueles usados para prever como as proteínas se dobram mostram uma forte correlação entre os locais de ligação minimamente frustrados e a especificidade do medicamento. O funil, uma representação visual da paisagem energética da proteína conforme ela se dobra, ajuda a localizar esses sites frustrados. Esses modelos podem levar a medicamentos mais bem elaborados e com menos efeitos colaterais. Crédito:Mingchen Chen / Rice University
p Modelos em escala atômica por cientistas da Rice University baseados naqueles usados para prever como as proteínas se dobram mostram uma forte correlação entre os locais de ligação minimamente frustrados e a especificidade do medicamento. O funil, uma representação visual da paisagem energética da proteína conforme ela se dobra, ajuda a localizar esses sites frustrados. Esses modelos podem levar a medicamentos mais bem elaborados e com menos efeitos colaterais. Crédito:Mingchen Chen / Rice University
p Saber exatamente onde as proteínas são frustradas pode contribuir muito para a fabricação de medicamentos melhores. p Esse é um dos resultados de um novo estudo realizado por cientistas da Rice University em busca dos mecanismos que estabilizam ou desestabilizam seções-chave de biomoléculas.
p Modelos em escala atômica do teórico de Rice Peter Wolynes, o principal autor e ex-aluno Mingchen Chen e seus colegas do Center for Theoretical Biological Physics mostram que não apenas algumas sequências específicas frustradas em proteínas são necessárias para permitir que funcionem, localizá-los também oferece pistas para obter uma melhor especificidade para medicamentos.
p Esse conhecimento também pode ajudar a desenvolver medicamentos com menos efeitos colaterais, Wolynes disse.
p O estudo de acesso aberto da equipe aparece em
Nature Communications .
p Os modelos em escala atômica enfocam as interações dentro de possíveis sítios de ligação, em vez da vasta maioria das interações em proteínas que orientam seu dobramento. Os modelos de resolução mais finos permitem a incorporação de cofatores como ligantes quimicamente ativos, incluindo moléculas de drogas. Os pesquisadores dizem que essa capacidade dá uma nova compreensão sobre por que os ligantes são mais bem capturados apenas por proteínas específicas e não por outras.
p "Ligantes não naturais, "também conhecido como drogas, tendem a se ligar melhor com aqueles bolsões frustrados em proteínas que se tornam minimamente frustrados uma vez que as drogas se ligam, Wolynes disse. Ter uma maneira de encontrar e aprender os detalhes desses sites minimamente frustrados ajudaria as empresas farmacêuticas a eliminar muitas tentativas e erros.
p "A maneira padrão de fazer design de drogas é experimentar 10, 000 sítios de ligação em uma proteína para encontrar aqueles que se encaixam, "Wolynes disse." Estamos dizendo que você não tem que amostrar todos os locais de ligação possíveis, apenas um número razoavelmente justo para entender as estatísticas do que poderia funcionar em ambientes locais.
p "É a diferença entre fazer uma votação e realmente ter uma eleição, "disse ele." A enquete é mais barata, mas você ainda precisará verificar as coisas. "
p Os pesquisadores do Rice são conhecidos por sua teoria da paisagem energética de como as proteínas se dobram. Geralmente emprega modelos de granulação grossa nos quais os aminoácidos são representados por apenas alguns locais.
p Essa estratégia exige menos poder de computação do que tentar determinar as posições ao longo do tempo de cada átomo em cada resíduo, e ainda assim provou ser altamente preciso em prever como as proteínas se dobram com base em suas sequências. Mas para este estudo, os pesquisadores modelaram proteínas e complexos de proteína-ligante em nível atômico para ver se conseguiam descobrir como a frustração dá a algumas partes de uma proteína a flexibilidade necessária para se ligar a outras moléculas.
p "Uma das melhores coisas sobre a modelagem da resolução de todos os átomos é que ela nos permite avaliar se as moléculas da droga se encaixam bem nos locais de ligação ou não, "Wolynes disse." Este método é capaz de mostrar rapidamente se um local de ligação para uma determinada droga será minimamente frustrado ou permanecerá uma região frustrada. Se depois que a molécula se liga o local permanece frustrado, a proteína pode se rearranjar ou a droga pode mudar sua orientação de tal forma que pode causar efeitos colaterais ”.
p Modelar os sites frustrados - e às vezes alterá-los para ver o que aconteceria - permite que os pesquisadores vejam como a especificidade do medicamento se correlaciona com os bolsões de ligação. Análise de frustração, eles escreveram, fornece "uma rota para a triagem de compostos mais específicos para a descoberta de drogas."
p "Este conceito de frustração estava lá no início do nosso trabalho sobre o dobramento de proteínas, "Wolynes disse." Quando o aplicamos a moléculas de proteínas reais, encontramos alguns exemplos em que o mecanismo de dobramento violou o que preveríamos de um funil perfeito. Então descobrimos que esses desvios da imagem do funil ocorreram onde a proteína estava, na verdade, um pouco frustrado.
p "Foi como a exceção que confirma a regra, "disse ele." Algo que é verdade o tempo todo pode ser trivial. Mas se não for verdade 1% das vezes, é um problema a ser resolvido, e conseguimos fazer isso com o AWSEM, nosso software de previsão de estrutura. "
p É possível estender o software para analisar a frustração no nível atômico, conforme descrito pelo grupo em outro artigo recente. Mas o custo computacional de rastrear cada átomo em uma proteína é tão alto que os pesquisadores precisavam encontrar uma maneira de amostrar os movimentos de regiões específicas onde a frustração pode confundir a rota de dobramento.
p "Mingchen percebeu que havia um algoritmo eficiente para amostrar os ambientes locais em sítios de ligação, mas manter a resolução atomística, "disse Wolynes, que notou ele e Chen, agora na indústria privada, estão usando os modelos para investigar possíveis terapêuticas, incluindo medicamentos relacionados com COVID-19.
