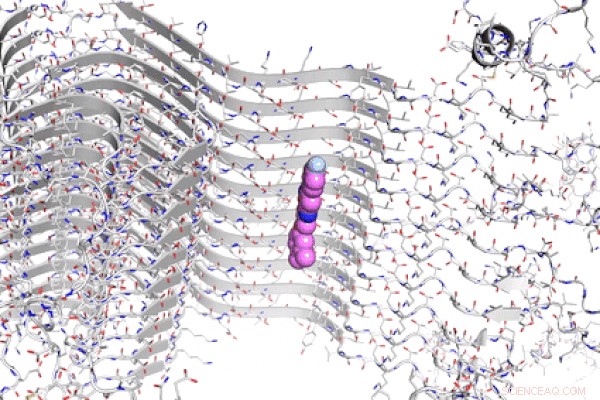
A estrutura química de uma fibrila de alfa-sinucleína com uma molécula "exemplar", mostrado como esferas coloridas, ligado a um local de ligação previamente identificado. Pesquisadores da Penn demonstraram recentemente um novo método para rastrear e identificar moléculas computacionalmente que podem ser desenvolvidas em sondas de imagem para estudar proteínas associadas à doença de Parkinson. Crédito:E. James Petersson
Para muitos dos 200, 000 pacientes com diagnóstico de doença de Parkinson nos Estados Unidos todos os anos, o diagnóstico geralmente ocorre apenas após o aparecimento de sintomas graves, como tremores ou dificuldades de fala. Com o objetivo de reconhecer e tratar doenças neurológicas precocemente, os pesquisadores estão procurando novas maneiras de criar imagens de moléculas biológicas que indiquem a progressão da doença antes que os sintomas apareçam. Um tal candidato, e uma marca conhecida da doença de Parkinson, é a formação de aglomerados de proteína alfa-sinucleína, e, enquanto esta proteína foi identificada há mais de 20 anos, uma maneira confiável de rastrear agregados de alfa-sinucleína no cérebro ainda não foi desenvolvida.
Agora, um novo estudo publicado em Ciência Química descreve uma abordagem inovadora para identificar moléculas que podem ajudar a rastrear a progressão da doença de Parkinson. Conduzido por pesquisadores nos laboratórios de E. James Petersson, Robert Mach, e Virginia Lee, este estudo de prova de conceito pode mudar o paradigma de como os pesquisadores rastreiam e testam novas moléculas para estudar uma ampla gama de doenças neurodegenerativas.
O estudo desses tipos de agregados de proteínas requer novos traçadores, moléculas radioativas que os médicos usam para criar imagens de tecidos e órgãos, para tomografia por emissão de pósitrons (PET). Como pesquisador sênior na área de desenvolvimento de rastreadores PET, Mach e seu grupo trabalharam por vários anos com a Michael J. Fox Foundation para desenvolver um traçador de alfa-sinucleína, mas, sem dados sobre a estrutura da proteína, eles não foram capazes de encontrar candidatos que fossem seletivos o suficiente para serem usados como uma ferramenta de diagnóstico.
Então, com a primeira publicação da estrutura da alfa-sinucleína e um aumento nas ferramentas disponíveis no campo da química computacional, Mach e Petersson começaram a colaborar no desenvolvimento de um traçador PET de alfa-sinucleína. Ao combinar seus respectivos conhecimentos em radioquímica e engenharia de proteínas, eles foram capazes de confirmar experimentalmente onde as moléculas traçadoras de potencial da proteína alfa-sinucleína foram capazes de se ligar, informações cruciais para ajudá-los a descobrir e projetar moléculas que seriam específicas para a alfa-sinucleína.
Em seu último estudo, os pesquisadores desenvolveram um método computacional de alto rendimento, permitindo que eles rastreiem milhões de moléculas candidatas, para ver quais se ligam aos locais de ligação conhecidos na alfa-sinucleína. Construindo um método publicado anteriormente, sua abordagem primeiro identifica um "exemplar, "uma pseudo-molécula que se encaixa perfeitamente no sítio de ligação da alfa-sinucleína. esse exemplo é comparado a moléculas reais que estão comercialmente disponíveis para ver quais têm uma estrutura semelhante. Os pesquisadores então usam outros programas de computador para ajudar a restringir a lista de candidatos para teste no laboratório.
Para avaliar o desempenho de seu método de triagem, os cientistas identificaram um pequeno subconjunto de 20 candidatos promissores dos 7 milhões de compostos que foram selecionados e descobriram que dois tinham afinidade de ligação extremamente alta para a alfa-sinucleína. Os pesquisadores também usaram tecidos cerebrais de camundongos fornecidos pelo grupo de Lee para validar ainda mais esse novo método. Os pesquisadores ficaram impressionados, e agradavelmente surpreso, por sua taxa de sucesso, que atribuem à natureza específica do seu método de pesquisa. "Certamente também há um pouco de sorte envolvida, "Petersson acrescenta, "Provavelmente, a maior surpresa é como funcionou bem."
A ideia de usar o método exemplar para resolver este problema veio ao primeiro autor e Ph.D. John "Jack" Ferrie se formou enquanto aprendia métodos de química computacional no Institute for Protein Design da University of Washington como parte de uma bolsa de verão da Parkinson's Foundation. "A bolsa de verão é projetada para treinar alunos em novos métodos que podem ser aplicados à pesquisa da doença de Parkinson, e isso é exatamente o que aconteceu aqui, "diz Petersson." As ideias que Jack trouxe formaram a base de um grande esforço em meu laboratório e no de Bob Mach para identificar rastreadores PET computacionalmente. "
Agora, como parte de um grande subsídio multi-institucional, Petersson, Mach, Lee, e muitos outros colaboradores estão prontos para aproveitar as lições aprendidas com essa descoberta para desenvolver rastreadores PET para Parkinson e outras doenças neurodegenerativas. "Eu realmente vejo isso como uma virada de jogo em como fazemos o desenvolvimento de sondas PET, "diz Mach." O significado é que somos capazes de rastrear milhões de compostos em um período muito curto de tempo, e somos capazes de identificar um grande número de compostos que provavelmente se ligarão com alta afinidade à alfa-sinucleína. Também vamos aplicar esse mesmo método ao desenvolvimento de outras sondas que são importantes, mas apresentam desafios para o campo. "
Ao desenvolver ferramentas confiáveis de alto rendimento que usam conhecimento detalhado da estrutura da proteína, o objetivo dos esforços futuros é encontrar novos candidatos a traçadores e levá-los à clínica assim que estiverem prontos para o teste. "Certamente está acelerado em comparação com o que é típico, "diz Petersson sobre os prazos da concessão." Isso pode levar de 10 a 15 anos na indústria, e estamos tentando fazer isso em cerca de cinco. "
Mach acrescenta que esse esforço é um exemplo perfeito de "como as coisas funcionam aqui na Penn, "com o sucesso possibilitado por colaborações entre pesquisadores com conjuntos de habilidades diversos e únicos." Penn é um ótimo lugar porque você tem muitas pessoas talentosas que têm um verdadeiro espírito de colaboração, e isso é o que é preciso para fazer ciência nos dias de hoje, " ele diz.