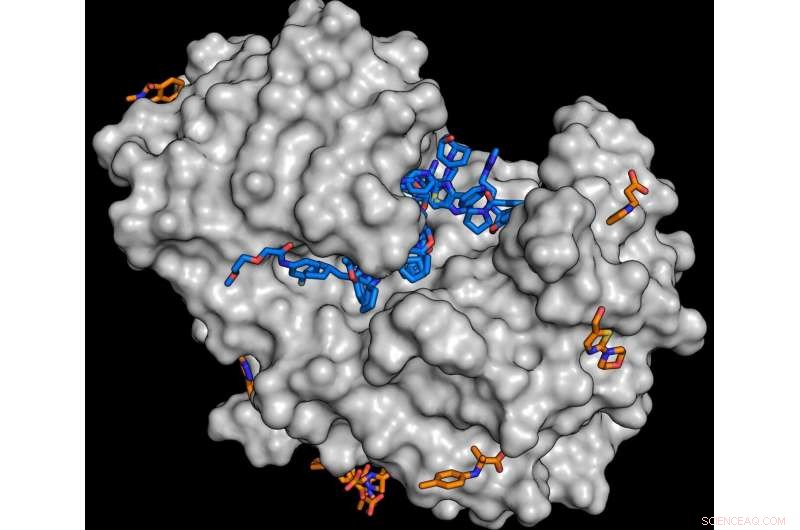
 p Para o estudo, a enzima endotiapepsina (cinza) foi combinada com moléculas da biblioteca de fragmentos. A análise mostra que várias substâncias são capazes de se acoplar à enzima (moléculas azuis e laranja). Cada substância encontrada é um ponto de partida potencial para o desenvolvimento de moléculas maiores. Crédito:J. Wollenhaupt / HZB
p Para o estudo, a enzima endotiapepsina (cinza) foi combinada com moléculas da biblioteca de fragmentos. A análise mostra que várias substâncias são capazes de se acoplar à enzima (moléculas azuis e laranja). Cada substância encontrada é um ponto de partida potencial para o desenvolvimento de moléculas maiores. Crédito:J. Wollenhaupt / HZB
p Para acelerar o desenvolvimento sistemático de medicamentos, a equipe MX do Helmholtz-Zentrum Berlin (HZB) e o Drug Design Group da Universidade de Marburg estabeleceram uma nova biblioteca de substâncias. Consiste em 1103 moléculas orgânicas que podem ser usadas como blocos de construção para novos medicamentos. A equipe MX já validou esta biblioteca em colaboração com o grupo FragMAX no MAX IV. A biblioteca de substâncias do HZB está disponível para pesquisas em todo o mundo e também desempenha um papel na pesquisa de substâncias ativas contra o SARS-CoV-19. p Para que os medicamentos sejam eficazes, eles geralmente precisam se encaixar nas proteínas de um organismo. Como uma chave em uma fechadura, parte da molécula do medicamento deve caber nos recessos ou cavidades da proteína-alvo. Por vários anos agora, a equipe do Departamento de Cristalografia Macromolecular (MX) do HZB liderada pelo Dr. Manfred Weiss juntamente com o Grupo de Design de Drogas liderado pelo Prof. Gerhard Klebe (Universidade de Marburg) tem, portanto, trabalhado na construção do que são conhecidas como bibliotecas de fragmentos. Estes consistem em pequenas moléculas orgânicas (fragmentos) com as quais as cavidades funcionalmente importantes na superfície das proteínas podem ser sondadas e mapeadas. Os cristais de proteína são saturados com os fragmentos e então analisados usando uma poderosa luz de raios-X. Isso permite que informações estruturais tridimensionais sejam obtidas em níveis de resolução atômica. Entre outras coisas, é possível descobrir o quão bem um fragmento de molécula específico se encaixa na proteína alvo. O desenvolvimento dessas bibliotecas de substâncias ocorreu como parte do projeto de pesquisa conjunto Frag4Lead e foi financiado pelo Ministério Federal Alemão de Educação e Pesquisa (BMBF).
p A equipe MX (MX significa Cristalografia Macromolecular) publicou agora o projeto de uma biblioteca de fragmentos quimicamente diversa chamada de biblioteca "F2X-Universal", que consiste em 1, 103 compostos. Uma seleção representativa de 96 compostos foi extraída desta biblioteca, que é conhecido como a tela de entrada F2X. Durante a publicação da biblioteca, esta seleção foi agora testada e validada com sucesso pela equipe MX do HZB na fonte de raios-X MAX IV em Lund, Suécia e em BESSY II.
p No estudo, as equipes de HZB e MAX IV verificaram a eficiência da biblioteca F2X Entry por meio da triagem de endotiapepsina e do complexo de proteína Aar2 / RnaseH como as enzimas-alvo. Na próxima etapa, a equipe MX usará toda a biblioteca universal.
p "Para o estudo atual, os especialistas em triagem de fragmentos no HZB — BESSY II trabalharam em estreita colaboração com a equipe do projeto FragMAX no MAX IV ", disse o Dr. Uwe Müller da equipe MX em HZB que ajudou a configurar as três linhas de luz MX em BESSY II, bem como a linha de luz BioMAX em MAX IV. "Isso permitiu que ambos os parceiros desenvolvessem suas próprias plataformas de tecnologia e as usassem para obter imagens das superfícies funcionais de diferentes proteínas. Essa será uma excelente base para uma colaboração futura entre MAX IV e HZB."
