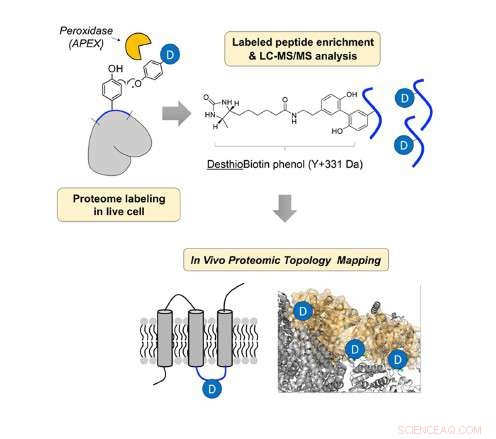
Identificação estrutural in vivo da proteína mitocondrial, usando peroxidase e destiobiotina-fenol. Crédito:UNIST
As proteínas de membrana constituem aproximadamente um quarto de todos os produtos genéticos e são o alvo de mais de 50 por cento de todos os medicamentos farmacêuticos modernos. O proteoma da membrana mitocondrial interna (IMM) desempenha um papel central na manutenção da fisiologia mitocondrial e do metabolismo celular. Apesar de sua importância, não houve nenhum método para revelar a topologia das proteínas da membrana mitocondrial em células vivas, até agora.
Um estudo recente, afiliado à UNIST desenvolveu uma nova técnica para entender as arquiteturas corretas de proteínas IMM, usando ferramentas químicas especiais. Ao resolver o estágio mais difícil de desenvolvimento de um novo medicamento, seu trabalho ajudará a acelerar o desenvolvimento de novas terapêuticas e curas.
Esta pesquisa foi liderada pela equipe do Professor Hyun-Woo Rhee de Química da UNIST em colaboração com o Professor Jong-Seo Kim no Centro do Centro de Pesquisa de RNA, dentro do Instituto de Ciências Básicas (IBS) da Universidade Nacional de Seul e Professor Jeong-Kon Seo da UNIST Central Research Facilities (UCRF). Os resultados do estudo foram publicados na edição de 15 de março do Jornal da American Chemical Society ( JACS )
IMM é um dos locais mais ativos para o metabolismo celular e está profundamente relacionado com várias doenças metabólicas humanas, incluindo câncer e doenças neurodegenerativas. Portanto, é crucial entender a arquitetura correta do proteoma IMM em células vivas para o desenvolvimento bem-sucedido e eficiente de terapêuticas direcionadas às mitocôndrias.
No estudo, O professor Lee e sua equipe de pesquisa revelaram a direção topológica in vivo de 135 proteínas IMM, usando uma sonda de radical destiobiotina-fenoxil gerada in situ com peroxidase geneticamente direcionada (APEX).
"A determinação da estrutura da proteína da membrana é uma das tarefas mais desafiadoras na análise da estrutura da proteína, "diz o professor Lee." Nossa identificação de informações estruturais sobre o proteoma da membrana mitocondrial interna pode fornecer informações valiosas para a arquitetura e conectoma do proteoma IMM em células vivas. "
A equipe de pesquisa projetou uma nova sonda química, destiobiotina-fenol e aplicou-o às proteínas IMM em células vivas. Então, eles identificaram a estrutura das proteínas de membrana por meio de espectrometria de massa (MS).
A peroxidase pode reagir com o peróxido de hidrogênio para formar o radical fenoxil. Então, o radical fenoxil pode reagir com o resíduo de tirosina na proteína proximal formando uma ligação covalente. No estudo, a equipe de pesquisa obteve as informações de topologia analisando o local rotulado de tirosina da proteína de membrana.
A maioria das análises de sequência de proteínas hoje usa espectrometria de massa (MS), que digere a amostra de proteína em peptídeos usando uma enzima apropriada. Análises anteriores, que usou ascorbato peroxidase geneticamente direcionado (APEX), não conseguiu resolver a identificação estrutural porque essas análises foram baseadas na detecção de peptídeos não marcados. Contudo, apenas o peptídeo marcado pode fornecer informações estruturais úteis, de acordo com a equipe de pesquisa.
Ao contrário das biomoléculas que são marcadas com biotina-fenol, proteínas e outros alvos marcados com destiobiotina-fenol podem ser eluídos sem agressividade, condições de desnaturação. Além disso, conforme o número de amostras de estrutura de proteína de membrana disponíveis obtidas via MS aumenta, a eficiência da identificação estrutural de proteínas de membrana também aumenta.
Devido ao curto tempo de vida dos radicais fenoxil gerados in situ por APEX direcionado submitocondrial e a impermeabilidade do IMM a moléculas pequenas, os resíduos de tirosina expostos a solvente de ambos os lados da matriz e do espaço intermembranar (IMS) das proteínas IMM foram marcados exclusivamente com a sonda radical em células vivas por Matrix-APEX e IMS-APEX, respectivamente e identificados por espectrometria de massa.
Por meio dessa análise, a equipe de pesquisa confirmou 58 topologias de proteínas IMM e determinou a direção topológica de 77 proteínas IMM cuja topologia no IMM não foi totalmente caracterizada.