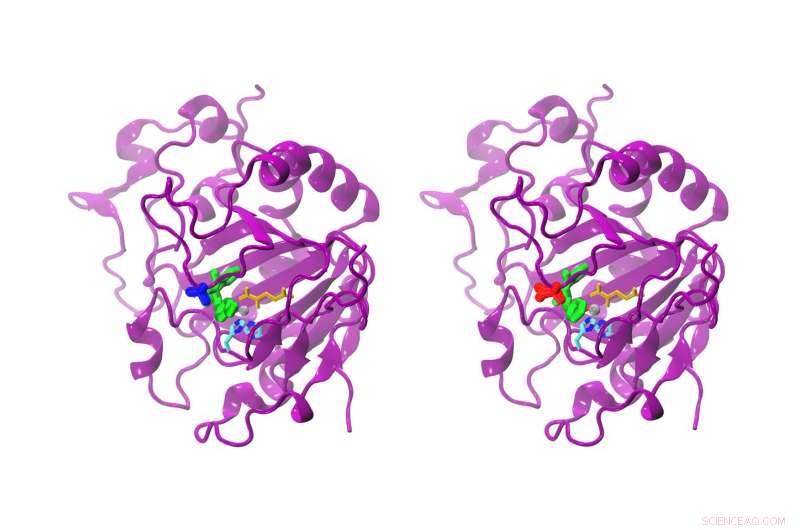
Embora a diferença entre o AsqJ natural e o modificado seja apenas um aminoácido, a reatividade é claramente diferente. O AsqJ natural (esquerda) possui uma valina na posição 72 (azul). A forma modificada possui uma isoleucina (vermelha) na posição 72. O substrato no sítio ativo é de cor verde, alfa-cetoglutarato amarelo, o átomo de ferro cinza, e duas cadeias de histidina ciano e azul. Crédito:Sophie Mader / TUM
Praticamente todos os processos bioquímicos envolvem enzimas que aceleram as reações químicas. Uma equipe de pesquisa da Universidade Técnica de Munique (TUM) decifrou pela primeira vez o mecanismo molecular da enzima AsqJ. Suas descobertas podem abrir novas opções na produção de moléculas farmaceuticamente ativas.
Sem enzimas, a natureza pararia. Essas minúsculas moléculas aceleram as reações bioquímicas ou as tornam possíveis em primeiro lugar. Mas como isso acontece em nível molecular? "Compreender a função exata das enzimas é um dos maiores desafios da bioquímica moderna, "diz Ville Kaila, Professor de Biocatálise Computacional na Universidade Técnica de Munique.
A equipe de pesquisa liderada por Ville Kaila e Michael Groll, Professor de Bioquímica da Universidade Técnica de Munique, tenho, pela primeira vez, decifrou o mecanismo da enzima aspoquinolona J (AsqJ), uma dioxigenase que ativa ligações de carbono com oxigênio.
A enzima AsqJ é particularmente excitante, pois catalisa uma cascata de reações químicas que levam à formação de compostos antibacterianos. Foi descoberto há apenas alguns anos no Aspergillus nidulans fungo. Os pesquisadores combinaram diferentes métodos para descobrir os segredos mantidos dentro da enzima:Primeiro, Alois Bräuer e o Prof. Michael Groll usaram a cristalografia de raios X para determinar a estrutura atômica tridimensional da molécula. Sophie Mader e Ville Kaila então usaram essas informações para realizar simulações de mecânica quântica em seus processos bioquímicos.
"Nossos cálculos ilustram como a enzima catalisa a formação do alcalóide quinolona, "relata Kaila." Pequenos detalhes têm efeitos surpreendentes:uma ligeira mudança no substrato, como a remoção de um pequeno grupo químico, é suficiente para praticamente parar a reação. "
Próximo, a equipe projetou computacionalmente uma nova variante da enzima que catalisa a formação de alcalóides quinolonas com o substrato modificado. Esta nova enzima foi produzida experimentalmente em bactérias e testada quanto à sua funcionalidade. "Os resultados foram impressionantes:a reação esperada ocorreu após apenas alguns segundos, "lembra Bräuer.
"Este experimento demonstra que nossa metodologia funciona e também é adequada para representar a funcionalidade de outras enzimas em nível molecular, "diz Ville Kaila. O design da enzima ainda está em um nível básico, mas tem um potencial enorme. No futuro, poderíamos ter como objetivo projetar medicamentos médicos computacionalmente, por exemplo.
"O trabalho demonstra que nossa metodologia é precisa e também adequada para estudar a funcionalidade de outras enzimas em nível molecular, "diz Ville Kaila. O projeto de enzimas ainda é uma pesquisa básica - mas tem um potencial enorme. Um objetivo de pesquisas futuras será projetar enzimas em um computador para, por exemplo, produzir novos medicamentos.