Estabelecer um mapa genético de genes bacterianos cruciais para a colonização de plantas por micróbios benéficos
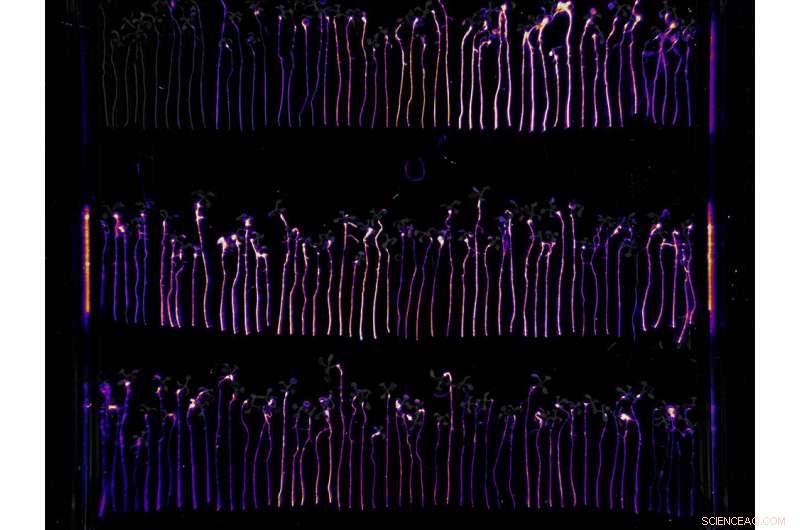 p Um ensaio bioluminescente ajudou os pesquisadores a quantificar visualmente a capacidade de colonização de cepas mutantes de P. simiae identificadas pela tela RB-TnSeq. Crédito:Benjamin Cole
p Um ensaio bioluminescente ajudou os pesquisadores a quantificar visualmente a capacidade de colonização de cepas mutantes de P. simiae identificadas pela tela RB-TnSeq. Crédito:Benjamin Cole
p Trabalhando com a bactéria promotora de crescimento vegetal Pseudomonas simiae, pesquisadores identificaram 115 genes que afetam negativamente sua capacidade de colonizar o sistema radicular de uma planta quando sofre mutação. p A saúde e o desenvolvimento de uma planta são influenciados pela complexa comunidade de micróbios que a rodeia. Ao identificar os genes bacterianos que podem alterar o quão bem os micróbios podem colonizar uma planta, os pesquisadores podem desenvolver abordagens direcionadas para melhorar a saúde e o crescimento das plantas para uma série de aplicações, incluindo maior rendimento de biomassa para produção de biocombustíveis.
p A saúde e o desenvolvimento de uma planta são influenciados por micróbios que residem dentro da planta (endófitos), no solo, e na estreita região onde as raízes das plantas interagem com o solo (rizosfera). Para entender melhor como os micróbios colonizam o ambiente radicular, pesquisadores do Joint Genome Institute, um DOE Office of Science User Facility, e seus colaboradores no Howard Hughes Medical Institute da University of North Carolina, aplicou uma abordagem de mutagênese de transposon de todo o genoma no modelo de bactéria promotora de crescimento de planta Pseudomonas simiae usando o modelo de planta Arabidopsis thaliana como hospedeiro para gerar um mapa de todo o genoma de genes bacterianos que afetam a eficácia da colonização microbiana.
p Por meio de sequenciamento de transposon com código de barras aleatoriamente (RB-TnSeq), a equipe identificou 115 genes que, quando mutado, têm capacidades de colonização de raiz reduzidas. Esses genes estão envolvidos em funções como o metabolismo do açúcar, síntese da parede celular, e motilidade. A equipe também identificou 243 genes que, quando mutado, alterar positivamente as capacidades de colonização de raízes, muitos deles provavelmente envolvidos no transporte e metabolismo de aminoácidos. Adicionalmente, a equipe identificou 43 genes aos quais muito pouca ou nenhuma informação funcional poderia ser atribuída. Os pesquisadores sugeriram que esses genes podem representar novas funções ou vias ainda a serem caracterizadas. O trabalho mostra que o RB-TnSeq pode ser aplicado para avaliar a colonização de raízes de plantas bacterianas in vivo.
p Entre os contribuintes deste projeto estava Sabah Ul-Hasan, um estagiário de 2015 através do DOE JGI / University of California, Programa de Estágio de Pós-Graduação Distinto da Merced Genomics. O programa oferece aos alunos de graduação da UC Merced experiência prática na pesquisa de ponta do genoma como parte do compromisso do DOE JGI em treinar a próxima geração de talentos científicos.
p Um dos principais desafios decorrentes do sequenciamento rápido é a atribuição de funções a novos genes. A abordagem RB-TnSeq usada aqui pode acelerar a associação de novos genes com características e comportamentos de importância para as missões DOE, como entender como os micróbios ajudam (ou atrapalham) o crescimento de safras que poderiam servir como matéria-prima de bioenergia. Alcançando os contribuintes genéticos fundamentais, como os 115 genes que regulam negativamente as interações micróbio-raiz da planta, ajudará a concentrar esforços futuros para o avanço desta pesquisa.
