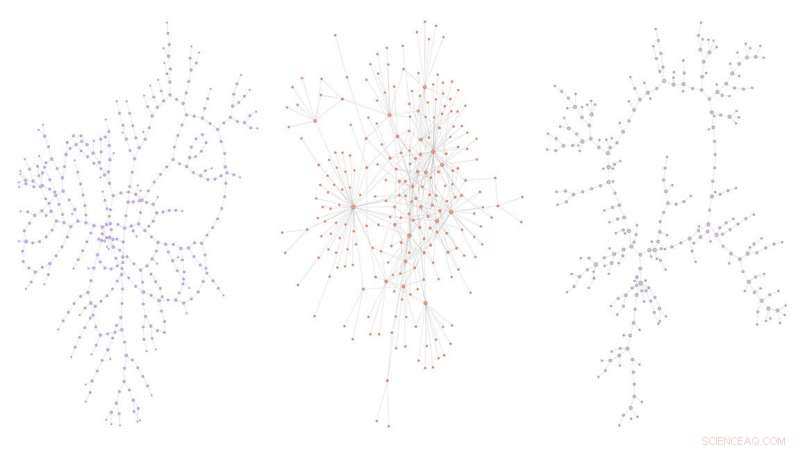
Um novo modelo matemático da estrutura das redes pode ajudar a encontrar novos medicamentos contra o câncer, acelerar o fluxo do tráfego e combater doenças sexualmente transmissíveis. Embora os três desafios pareçam diversos, todos eles poderiam se beneficiar de uma teoria que ajuda a descobrir informações sobre uma rede, analisando sua estrutura. Algoritmos de previsão de link bem-sucedidos já existem para certos tipos de redes, mas os pesquisadores analisaram redes estruturadas de forma diferente para chegar a seu algoritmo alternativo. Esta imagem mostra três redes semelhantes a árvores:redes de distribuição metropolitana de água (da esquerda para a direita), Comunicação no Twitter e contatos sexuais. Crédito:Keke Shang
Um novo modelo matemático da estrutura das redes pode ajudar a encontrar novos medicamentos contra o câncer, acelerar o fluxo do tráfego e combater doenças sexualmente transmissíveis.
Embora os três desafios pareçam diversos, todos eles poderiam se beneficiar de uma teoria que ajuda a descobrir informações desconhecidas sobre uma rede, analisando sua estrutura. O estudo foi publicado na revista Caos .
Um exemplo de como preencher os links que faltam pode ser útil é na escolha de um alvo genético para um medicamento contra o câncer, disse o membro da equipe Michael Small, da University of Western Australia.
"Digamos que você tenha uma rede de genes que estão de alguma forma conectados, e existem alguns alvos de drogas conhecidos. Mas se você não conhece todos os genes, você gostaria de fazer suposições sobre as informações que você não precisa descobrir quais podem ser outros alvos prováveis para investigar, "Small disse.
Algoritmos de previsão de link bem-sucedidos já existem para certos tipos de redes, mas os pesquisadores analisaram redes estruturadas de forma diferente para chegar a seu algoritmo alternativo.
Um exemplo de previsão de link tradicional pode ser uma rede social sugerindo amigos com os quais se conectar. Duas pessoas que compartilham um amigo provavelmente são amigas com uma conexão direta. Adicionar esta terceira conexão cria um triângulo, que é uma estrutura onde os algoritmos existentes se concentram.
Em contraste, os autores se concentraram em redes semelhantes a árvores, que têm muitos ramos, mas muito poucas ligações cruzadas entre os ramos. Eles estudaram três conjuntos de dados de exemplo:a rede social Twitter, uma rede de distribuição de água e uma rede de contato sexual.
Eles descobriram que essas três redes semelhantes a árvores podem ser caracterizadas por uma série de parâmetros, como a distância média entre pontos de ramificação na rede, o tamanho dos loops e uma comparação do número de links que os nós adjacentes têm - uma medida da regularidade da rede que descreve a heterogeneidade.
Os autores desenvolveram então um algoritmo que sugeria links que preservariam essas características da rede.
Eles testaram essa abordagem pegando uma rede conhecida e removendo links dela e vendo se o algoritmo poderia prever onde os links ausentes deveriam estar.
A equipe descobriu que o algoritmo teve um desempenho melhor (cerca de 44% para a rede do Twitter, cerca de 15% para a rede de contato sexual e cerca de 4% para a rede de distribuição de água) para redes semelhantes a árvores do que a maioria dos algoritmos convencionais que dependem de outros parâmetros, como presumir que indivíduos altamente conectados atrairão mais conexões (conhecido como apego preferencial) ou construir muitas conexões triangulares (conhecido como agrupamento).
O autor Keke Shang atribui o sucesso da equipe a pensar em exemplos de redes do mundo real.
"Espero que possamos fazer com que a tecnologia de rede sirva melhor às nossas vidas, " ele disse.