Crédito:Universidade da Pensilvânia
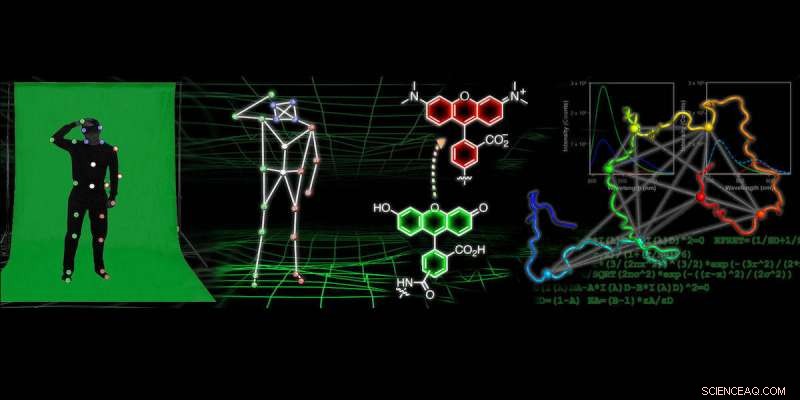
Em muitos filmes de animação modernos, o truque para obter movimentos realistas para personagens e objetos individuais está na tecnologia de captura de movimento. Este processo muitas vezes envolve alguém vestindo um traje de rastreamento coberto de pequenas, bolas coloridas enquanto uma câmera captura a posição dessas bolas coloridas, que é então usado para representar como a pessoa está se movendo.
Pesquisadores da Universidade da Pensilvânia estão desenvolvendo uma tecnologia semelhante para obter "filmes" de resolução atômica que rastreiam como as proteínas se dobram e mudam de forma. Para gerar esses filmes, os cientistas rotulam a proteína com sondas em várias posições e observam o movimento dessas etiquetas. Os dados de fluorescência nas posições relativas das sondas podem então ser usados para construir modelos computacionais da estrutura da proteína em detalhes atômicos. Esta pesquisa pode levar a melhorias nos medicamentos usados para tratar doenças neurodegenerativas, bem como novos métodos de geração de imagens que podem levar à sua detecção antecipada.
A pesquisa foi um esforço multidisciplinar liderado por E. James Petersson. O artigo foi publicado em Biophysical Journal .
"Uma das grandes questões fundamentais da bioquímica é como as proteínas se dobram em uma determinada forma, "disse Petersson, "e isso é ditado pela sequência de aminoácidos na proteína. As informações em todas as interações das cadeias laterais de aminoácidos de alguma forma levam a que se dobrem em uma forma adequada."
Em cenários saudáveis, Petersson disse, que a forma adequada permite que a proteína tenha funções diferentes, como o transporte de oxigênio no sangue ou a transformação em "máquinas moleculares" que, em última análise, levam aos movimentos musculares, como os necessários para caminhar e correr.
Mas em certos estados de doença, particularmente em doenças neurodegenerativas, como Alzheimer e Parkinson, as proteínas desdobram-se em uma forma doentia, o que pode fazer com que várias cópias das proteínas se agreguem em "emaranhados semelhantes a espaguete ou fibrilas longas". Essas fibrilas, Petersson disse, são tóxicos para os neurônios, que está por trás das doenças de Alzheimer e Parkinson.
"Uma vez que tudo isso envolve o enovelamento de proteínas em uma determinada forma, então o que gostaríamos de fazer é rastrear as mudanças na forma das proteínas, "disse ele." Há uma série de técnicas diferentes que podem ser usadas para fazer isso, mas gostamos da fluorescência porque você pode adquirir dados de fluorescência com rapidez suficiente para realmente ver as proteínas dobrarem em tempo real. Em última análise, gostaríamos de tentar observar o enovelamento das proteínas nas células. "
Para obter informações sobre a forma da proteína usando sondas fluorescentes, pesquisadores usam uma técnica chamada transferência de energia de ressonância fluorescente, o que requer que eles meçam muitas distâncias entre diferentes pontos na proteína e, em seguida, usem essa informação para entender sua forma, semelhante à tecnologia de captura de movimento.
Nesse artigo, os pesquisadores fizeram cerca de 30 medições de diferentes distâncias dentro da proteína alfa-sinucleína em diferentes estados onde ela está mudando de forma. Eles então usaram essa coleção de medidas de distância em combinação com modelagem computacional complexa para obter estruturas de resolução atômica da forma da proteína.
Ferrie recebeu uma bolsa da Fundação da Doença de Parkinson para passar o verão no laboratório de David Baker na Universidade de Washington, onde Rosetta, um dos programas mais comumente usados para modelar proteínas, foi desenvolvido.
"Rosetta é projetado para modelar proteínas bem dobradas estáveis, "Petersson disse, "não proteínas desordenadas que podem mudar de forma, então Jack teve que reescrever muito o código para poder modelar essas proteínas indisciplinadas. "
Ferrie e Yoon fizeram uma série de medições experimentais que poderiam ser usadas para direcionar o enovelamento da proteína para ser consistente com as medições experimentais, o que lhe permitiu modelar a forma da proteína.
Por ser uma nova abordagem, os pesquisadores queriam demonstrar que as estruturas que saíam dos modelos computacionais eram consistentes com a realidade. Para fazer isso, eles conduziram três tipos de experimentos para combinar dados reais com os modelos que saíram desses experimentos de fluorescência.
Eles colaboraram com o grupo de Rhoades para validar as estruturas modeladas usando medições de fluorescência de molécula única feitas por Pan. Trabalhando no grupo de Fakhraii, Lin, usou um tipo diferente de técnica chamada microscopia de força atômica para criar imagens da proteína e validar os modelos. Chris Dobson, um professor de química na Universidade de Cambridge, compartilharam dados de ressonância magnética nuclear sobre alfa-sinucleína que forneceram uma confirmação adicional da precisão dos modelos de computador.
O artigo representa uma das maiores bibliotecas de proteínas marcadas com fluoróforos sintéticos já relatadas. De acordo com Petersson, os pesquisadores precisaram fazer muitas medições de distância em diferentes regiões da proteína para ter dados suficientes para gerar modelos computacionais. Ferrie e Haney tiveram que criar uma abordagem simplificada para conectar diferentes conjuntos de sondas que funcionassem em diferentes faixas de distância.
Os pesquisadores agora estão trabalhando para aplicar esta técnica para modelar a estrutura da proteína nas formas agregadas que são tóxicas para os neurônios e para modelar sua resposta a drogas que fariam com que ela mudasse de forma, evitando esta agregação.
"A capacidade de observar uma proteína conforme ela muda de forma, "disse Petersson, "e obter estruturas a partir disso é uma meta científica básica muito importante para a qual temos trabalhado por 10 anos. Houve alguns avanços muito impressionantes na obtenção de estruturas de proteínas em doenças neurodegenerativas, mas a técnica de fluorescência tem potencial para fazer isso em células vivas, que nenhuma outra técnica tem a capacidade de fazer. "
De acordo com Petersson, proteínas que adotam formas múltiplas, empilhar cópias diferentes umas sobre as outras e agregar, como fazem em doenças neurodegenerativas, são um problema bioquímico básico importante que outras técnicas de biologia estrutural não podem realmente resolver. A better understanding of how that works and what those shapes are has the potential to make an impact on diseases such as Parkinson's and to give researchers an opportunity to figure out how drug or diagnostic models interact with the protein.
"We're working on being able to generate model structures that actually show what is the effect of these drugs, " Petersson said. "We take the protein with the fluorescent labels, add the drug, allow the protein to change shape, make fluorescence measurements and then take those back to the computational modeling so we can actually see the structural effect of these drugs. Hopefully this will lead to more of a rational understanding so that better second and third generation drugs can be made."
The researchers are also collaborating with Robert Mach, the Britton Chance Professor of Radiology in Penn's Perelman School of Medicine. Mach's group is interested in developing positron emission tomography imaging probes that can be used to bind to the aggregated forms of proteins and image them in patients.
"There are some promising drugs for treating neurodegenerative diseases such as Alzheimer's and Parkinson's, that could block this formation of aggregates, " Petersson said, "but the problem is that, by the time people show cognitive or motor-tremor symptoms, it's too late to use these drugs because there's already too much neurodegeneration. If you're getting aggregates in your brain, even if you're not showing any behavioral changes or learning deficits, these probes could noninvasively image the aggregates. By achieving a rational understanding of what the protein structure is, we hope we can help with that work moving forward."