Os pesquisadores usam nano-pinças baseadas em DNA para medir as forças entre os nucleossomos
 p Crédito:Chris Hohmann (NIM); Dietz Lab (TUM)
p Crédito:Chris Hohmann (NIM); Dietz Lab (TUM)
p O modo de empacotamento do DNA genômico no núcleo da célula determina os padrões de expressão gênica. Pesquisadores de Munique usaram nano-pinças baseadas em DNA para medir as forças entre os nucleossomos, as unidades básicas de embalagem do DNA nuclear. p Cada célula humana contém cerca de dois metros de ácido desoxirribonucléico (DNA), que codifica a informação genética que especifica as estruturas e funções celulares. Além disso, este DNA "genômico" é compactado no núcleo da célula, que tem menos de 10 micrômetros de diâmetro. Isso significa que o DNA nuclear deve ser embalado, principalmente pela interação com proteínas específicas. A unidade de embalagem básica é uma partícula feita de proteínas chamadas histonas, em torno do qual o DNA é envolvido. Comparável a pequenos carretéis, essas estruturas são chamadas de nucleossomos. Os nucleossomos, por sua vez, estão ligados uns aos outros por segmentos de DNA que se estendem entre as partículas do núcleo e não estão envolvidos em torno delas. Visto sob o microscópio eletrônico, o DNA empacotado em nucleossomos se assemelha a contas em um fio.
p O próximo nível de embalagem envolve a interação mútua de nucleossomos, e as estruturas de ordem superior resultantes ainda não foram completamente caracterizadas. Uma equipe de cientistas liderada por Hendrik Dietz da Universidade Técnica de Munique e Philipp Korber do Centro Biomédico da LMU deu um passo substancial para resolver este enigma:pela primeira vez na história, eles conseguiram medir diretamente as forças de atração que atuam entre os nucleossomos. Seus resultados aparecem nas revistas
Avanços da Ciência e
Nano Letras .
p
Origami de DNA:integrando nucleossomos nas pinças
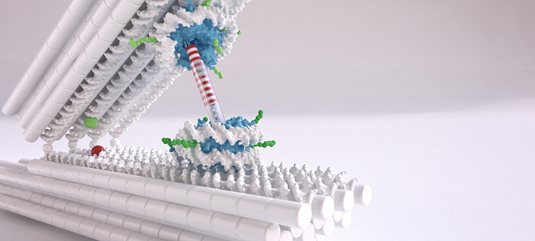
p Dietz, titular da cadeira de Biofísica Experimental da TUM, usa DNA como material de construção para construir estruturas moleculares - uma tecnologia conhecida como origami de DNA. Ele e sua equipe agora usaram o método para criar estruturas que consistem em duas barras de DNA rígidas conectadas por uma junta flexível que atua como uma mola. Eles podem ser usados como pinças para medir a força das interações entre os nucleossomos. Um nucleossomo é conectado a cada braço da pinça. "Podemos controlar a posição e orientação dos nucleossomos nas pinças de DNA com um alto grau de precisão, "diz Dietz." Isso é muito importante quando se trata de realmente ser capaz de medir as interações. "
p Os pesquisadores do LMU assumiram a tarefa de desenvolver estruturas de nucleossomos que podem ser integradas às pinças. Philipp Korber, Privatdozent e líder de grupo na cadeira de Biologia Molecular do BMC, explica:"Normalmente, as duas extremidades de fita dupla do DNA enroladas em torno do nucleossomo estão muito próximas uma da outra. Mas o que precisávamos eram duas fitas simples salientes, mais perto do meio. Este foi um problema significativo, uma vez que tal configuração pode desestabilizar toda a estrutura. Mesmo assim, a integrante da nossa equipe, Corinna Lieleg, conseguiu encontrar os locais certos para essas alças. "
p Os pesquisadores conseguiram medir uma interação muito fraca entre nucleossomos integrados, equivalente a uma força atrativa de 1,6 kcal / mol, em uma faixa de cerca de 6 nanômetros (nm). As orientações dos nucleossomos entre si dificilmente surtiram efeito. Contudo, modificações químicas particulares nas proteínas histonas enfraqueceram ainda mais as interações.
p
O problema da fibra de 30 nm
p O resultado pode ajudar a resolver uma disputa científica atual. De acordo com a teoria atual, os nucleossomos formam um tipo de superespiral com diâmetro de 30 nanômetros, a chamada fibra de 30 nm. Até aqui, Contudo, esta estrutura de 30 nm de ordem superior nunca foi observada em células vivas. Ainda é altamente controverso se a cromatina realmente assume a forma de uma superespiral. De fato, as diminutas forças de atração entre os nucleossomos, que os pesquisadores já mediram com sucesso, parecem contradizer a teoria. "Nossos dados apontam para estruturas muito suaves que são facilmente deformadas por influências externas, "diz Dietz.
p Como os nucleossomos são organizados em estruturas de ordem superior é uma questão fundamentalmente importante, pois tem implicações profundas para o controle da expressão gênica. Apenas aqueles genes que se encontram dentro da cromatina relativamente não compacta são acessíveis para 'ativação', que permite que as proteínas que codificam sejam produzidas pela maquinaria celular.
p
A regulação gênica por meio do empacotamento de DNA dá errado nas células cancerosas
p "Nos últimos dez anos, tornou-se claro que muitas das mudanças e mutações que transformam as células em células cancerosas ocorrem neste nível, "Korber diz. Em uma célula cancerosa, os mecanismos normais que determinam quais genes estão ativos e quais são inativos são interrompidos. As regiões genômicas que não deveriam ser acessíveis são deixadas em aberto e vice-versa. "Contudo, se apenas a embalagem estiver com defeito, e não o próprio gene, deve, em princípio, ser possível restaurar a embalagem adequada novamente. "
p Os pesquisadores planejam usar a técnica de pinça molecular para investigar outras estruturas. "Em biologia, a orientação das estruturas em relação umas às outras é sempre importante, "diz Korber." Agora temos um tipo de grampo molecular que podemos usar para controlar especificamente a orientação espacial das estruturas entre si. "
