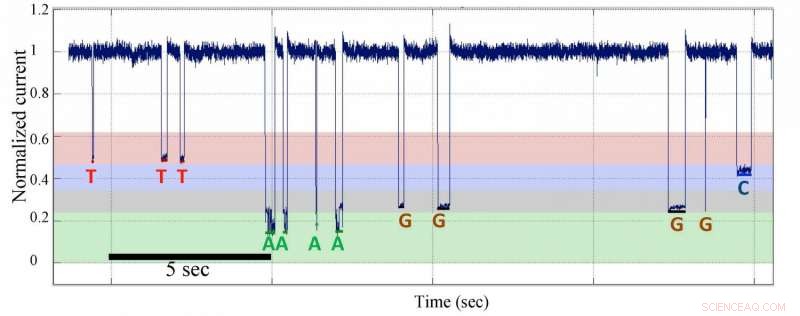
 p Sequenciamento de DNA nanoporo de molécula única por dados de síntese de um modelo com sequências de homopolímero. Crédito:Jingyue Ju, Columbia Engineering
p Sequenciamento de DNA nanoporo de molécula única por dados de síntese de um modelo com sequências de homopolímero. Crédito:Jingyue Ju, Columbia Engineering
p Pesquisadores da Universidade de Columbia, com colegas da Genia Technologies (Roche), A Universidade de Harvard e o Instituto Nacional de Padrões e Tecnologia (NIST) relatam a obtenção de sequenciamento eletrônico de DNA de molécula única em tempo real com resolução de base única usando uma matriz de nanoporos de proteína. p O sequenciamento de DNA é a tecnologia-chave para iniciativas de medicina personalizada e de precisão, permitindo descobertas rápidas na ciência biomédica. A sequência completa do genoma de um indivíduo fornece marcadores e diretrizes importantes para diagnósticos médicos, cuidados de saúde, e manter uma vida saudável. A data, o custo e a velocidade envolvidos na obtenção de sequências de DNA altamente precisas têm sido um grande desafio. Embora vários avanços tenham sido feitos na última década, os instrumentos de sequenciamento de alto rendimento amplamente usados hoje dependem da ótica para a detecção de quatro blocos de construção de DNA:A, C, G e T. Para explorar recursos de medição alternativos, O sequenciamento eletrônico de um conjunto de modelos de DNA foi desenvolvido para análise genética. Sequenciamento de filamentos de nanopore, em que um DNA de fita simples é enfiado através dos poros em nanoescala sob uma voltagem elétrica aplicada para produzir sinais eletrônicos para determinação de sequência em nível de molécula única, foi desenvolvido recentemente; Contudo, porque os quatro nucleotídeos são muito semelhantes em suas estruturas químicas, eles não podem ser facilmente distinguidos usando este método. Os pesquisadores estão, portanto, buscando ativamente a pesquisa e o desenvolvimento de uma plataforma de sequenciamento eletrônico de DNA de uma única molécula, pois ela tem o potencial de produzir um sequenciador de DNA miniaturizado capaz de decifrar o genoma para facilitar a medicina de precisão personalizada.
p Uma equipe de pesquisadores da Columbia Engineering, chefiado por Jingyue Ju (Samuel Ruben-Peter G. Viele Professor de Engenharia, Professor de Engenharia Química e Farmacologia, Diretor do Centro de Tecnologia de Genoma e Engenharia Biomolecular), com colegas da Harvard Medical School, liderado por George Church (professor de genética); Genia Technologies, liderado por Stefan Roever (CEO da Genia); e John Kasianowicz, o investigador principal do NIST, desenvolveram um sistema completo para sequenciar DNA em nanoporos eletronicamente em nível de molécula única com resolução de base única. Este trabalho, intitulado, "Sequenciamento Eletrônico de DNA de Molécula Única em Tempo Real por Síntese Usando Nucleotídeos Marcados com Polímero em uma Matriz Nanopore, "é publicado na revista,
Proceedings of the National Academy of Sciences (
PNAS ) Edição antecipada.
p Anteriormente, pesquisadores dos laboratórios de Ju em Columbia e Kasianowicz no NIST relataram o princípio geral de sequenciamento de nanopore por síntese (SBS), a viabilidade de projeto e síntese de nucleotídeos marcados com polímero como substratos para a DNA polimerase, a detecção e a diferenciação dos marcadores de polímero por nanopore no nível de molécula única [
Relatórios Científicos 2, 684 (2012) DOI:10.1038 / srep00684]. O actual
PNAS artigo descreve a construção do sistema nanopore SBS completo para produzir dados de sequenciamento eletrônico de molécula única com resolução de base única. Esta estratégia SBS distingue com precisão quatro bases de DNA detectando e diferenciando eletronicamente quatro marcadores de polímero diferentes ligados ao 5'-fosfato dos nucleotídeos durante sua incorporação em uma fita de DNA em crescimento catalisada pela polimerase, uma enzima sintetizadora de DNA. Os pesquisadores projetaram e sintetizaram novos nucleotídeos marcados no fosfato terminal com polímeros baseados em oligonucleotídeos para realizar SBS nanopore em uma plataforma de arranjo de nanoporos de proteína α-hemolisina. As marcas nos nucleotídeos marcados com polímero, que foram verificados como substratos ativos para a DNA polimerase, produzir diferentes níveis de bloqueio de corrente elétrica. Eles construíram uma matriz de nanopore em um chip eletrônico contendo vários eletrodos; a matriz é composta de canais de proteína que foram acoplados a uma DNA polimerase que foi ligada a um molde de DNA preparado. A adição de nucleotídeos marcados com polímeros projetados de maneira personalizada à matriz de nanoporos ativa a síntese de DNA. Ao bloquear a corrente iônica do canal em diferentes níveis, as tags distintas fornecem uma leitura da sequência do modelo em tempo real com resolução de base única.
p Como Carl Fuller, autor principal, Cientista Pesquisador Sênior Adjunto do Laboratório Ju do Departamento de Engenharia Química da Columbia e Diretor de Química da Genia, aponta, "A novidade de nossa abordagem SBS nanopore começa com o design, síntese, e seleção de quatro nucleotídeos marcados com polímero diferentes. Usamos uma DNA polimerase covalentemente ligada ao nanopore e os nucleotídeos marcados para realizar SBS. Durante a replicação do DNA ligado à polimerase, a etiqueta de cada nucleotídeo complementar é capturada no poro para produzir um sinal elétrico único. Quatro marcações de polímero distintas produzindo assinaturas distintas que são reconhecidas pelo detector eletrônico no chip de matriz nanopore são usadas para determinação de sequência. Assim, As sequências de DNA são obtidas para muitas moléculas únicas em paralelo e em tempo real. As quatro etiquetas de polímero são projetadas para oferecer distinções muito melhores entre si, em contraste com as pequenas diferenças entre os quatro nucleotídeos de DNA nativos, superando assim o grande desafio enfrentado por outros métodos de sequenciamento nanopore direto. "Além disso, as tags podem ser otimizadas em relação ao tamanho, cobrar, e estrutura para fornecer resolução ideal no sistema SBS nanopore.
p "Este projeto emocionante reúne cientistas e engenheiros da academia e da indústria com experiência combinada em engenharia molecular, nanotecnologia, genômica, eletrônica e ciência de dados para produzir revolucionário, plataformas de diagnóstico genético econômicas com potencial sem precedentes para medicina de precisão, "diz Ju." Estamos extremamente gratos pelo generoso apoio do NIH que nos permitiu fazer um rápido progresso na pesquisa e desenvolvimento da tecnologia nanopore SBS, e as contribuições pendentes de todos os membros do nosso consórcio de pesquisa. "
p De acordo com Ju, os pesquisadores já foram além do que foi demonstrado no
PNAS estudo onde os dados de sequenciamento foram obtidos em um sequenciador de protótipo inicial baseado em nanopore SBS. O rendimento e o desempenho do sequenciador atual progrediram além do que foi relatado no
PNAS papel. A viabilidade de alcançar comprimentos de leitura de mais de 1000 bases de DNA foi recentemente alcançada. Daqui para frente, a equipe de pesquisa colaborativa continuará a otimizar as tags ajustando os linkers, estrutura, e carga no nível molecular, e o ajuste fino da polimerase e da eletrônica para o sistema nanopore SBS com o objetivo de sequenciar com precisão um genoma humano inteiro rapidamente e a baixo custo, permitindo assim que seja usado em diagnósticos médicos de rotina.
