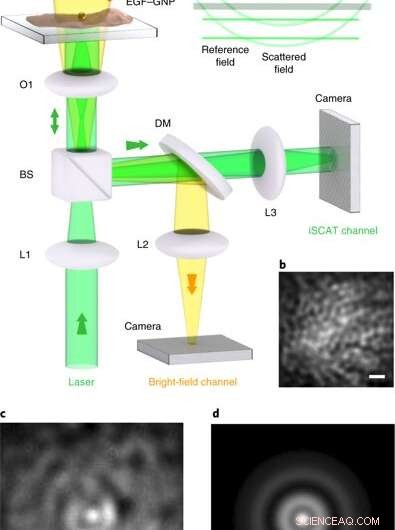
Microscopia iSCAT em células vivas. uma, Arranjo experimental do microscópio iSCAT para imagens de células vivas. As células são banhadas em um prato com fundo de vidro em meio Leibowitz. (a) a micropipeta entrega as sondas EGF-GNP diretamente na cultura de células, onde eles têm como alvo específico a proteína EGFR na membrana celular. O canal de iluminação de campo claro de cima auxilia na inspeção da cultura, mas não é necessário para imagens iSCAT. L1 – L3, lentes; O1, × 100 objetivo; BS, Divisor de feixe 90:10; DM, Espelho dicróico de passagem curta de 590 nm. A imagem iSCAT foi realizada com intensidades de iluminação de 1–8 kW cm − 2, que são conhecidos por serem viáveis para HeLa no comprimento de onda de interesse. Inserir, as frentes de onda dos campos que contribuem para o sinal iSCAT. (b), Uma seção da membrana da célula HeLa antes da rotulagem, visto via reflexão iSCAT. (c), Imagem iSCAT da membrana celular, incluindo uma sonda EGF-GNP ligada. (d), O PSF extraído de c. Barras de escala em b – d são 1 μm. Crédito: Nature Photonics , doi:10.1038 / s41566-019-0414-6
As funções celulares são ditadas pelo intrincado movimento das proteínas nas membranas que se estendem por uma escala de nanômetros a micrômetros, dentro de um intervalo de tempo de microssegundos a minutos. Contudo, este rico parâmetro de espaço é inacessível usando microscopia de fluorescência, embora esteja ao alcance do rastreamento de partículas de espalhamento interferométrico (iSCAT). A nova técnica iSCAT é, Contudo, altamente sensível a proteínas únicas e não marcadas, causando assim a coloração de fundo não específica como um desafio substancial durante a imagem celular.
Em um estudo recente, Richard W. Taylor e colegas dos departamentos interdisciplinares de Física e Biologia da Alemanha desenvolveram uma nova abordagem de processamento de imagens para superar essa dificuldade. Eles usaram o método para rastrear o receptor do fator de crescimento epidérmico transmembrana (EGFR) com precisão em escala nanométrica em três dimensões (3-D). A técnica permitiu a geração de imagens de microssegundos a minutos. Os cientistas forneceram exemplos de movimento e confinamento em nanoescala usando o método para processos ubíquos de imagem, como difusão em membranas plasmáticas, transporte em filópodes e movimento rotacional durante a endocitose. Os resultados agora são publicados em Nature Photonics .
Embora o progresso constante na microscopia de fluorescência tenha permitido aos cientistas monitorar eventos celulares em escala nanométrica, ainda há muito a ser realizado com sistemas de imagem avançados. Os desafios da microscopia de fluorescência ocorreram devido à taxa de emissão finita de uma fonte fluorescente (molécula de corante ou ponto quântico semicondutor), onde poucas emissões de fótons durante um período de tempo muito pequeno impediram a geração de imagens eficazes ou prolongadas. A dificuldade central da microscopia baseada em espalhamento é relativa à sonda nanoscópica, que compete contra o ruído de fundo e uma baixa relação sinal-ruído (SNR); limitar o potencial de geração de imagens a apenas alguns nanômetros em experimentos de rastreamento de alta velocidade.

Vídeo bruto de uma nanopartícula de ouro-fator de crescimento epidérmico (EGFR – GNP) difundindo-se em uma membrana celular HeLa. Crédito: Nature Photonics , doi:10.1038 / s41566-019-0414-6
No presente trabalho, Taylor et al. usaram microscopia de espalhamento interferométrico (iSCAT) para rastrear proteínas em membranas de células vivas. O método pode visualizar as interações sonda-célula para entender a dinâmica entre a difusão e a topologia local. Durante os experimentos, os cientistas usaram nanopartículas de ouro (GNPs) para rotular os receptores do fator de crescimento epidérmico (EGFRs) em células HeLa. Os EGFRs são proteínas transmembrana do tipo I que podem detectar e responder a sinais extracelulares, cuja sinalização aberrante está ligada a uma variedade de doenças. Taylor et al. mostraram a proteína marcada com GNP como um 'nano-rover' que mapeou a nano-topologia de características celulares, como terrenos de membrana, filopódios e estruturas de clatrina. Eles forneceram exemplos de subdifusão e movimento de confinamento nanoscópico de uma proteína em 3-D em alta resolução temporal e longos pontos de tempo.
Nos experimentos, Taylor et al. introduziu as sondas do fator de crescimento epidérmico-nanopartícula de ouro (EGF-GNP) na câmara de amostra do microscópio usando uma micropipeta para marcar os EGFRs (receptores do fator de crescimento epidérmico) nas células HeLa e verificou que as sondas estimularam os EGFRs. Estudos anteriores já haviam indicado que o tamanho da sonda poderia influenciar as taxas de difusão de lipídios em membranas sintéticas, embora não afetem o modo de difusão. Adicionalmente, em células vivas, o apinhamento molecular foi desprezível para partículas iguais ou menores que 50 nm.
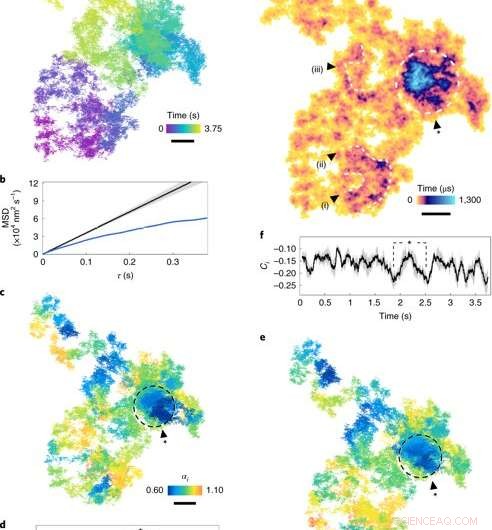
Difusão na membrana plasmática. (uma), Uma trajetória difusional lateral (tempo de exposição de 17,5 μs, veja a escala de cores para cronologia). (b), MSD (deslocamento quadrático médio) versus τ. A curva azul mostra o MSD de a. A curva preta é a difusão normal simulada (α =1), com o envelope cinza indicando a incerteza. (c), O expoente difusional de janelas rolantes (escala de cores) ao longo da trajetória. As regiões de subdifusão (α <1) são indicadas por tons mais escuros. (d), αi através do tempo. O sombreado cinza representa uma incerteza média de 7 ± 4%, correspondendo a um intervalo de confiança de 95% para uma janela de 100 ms (1, 000 quadros) e τ =250 μs. Os pontos marcados com o asterisco correspondem ao círculo em c. (e), A direção de passo Ci para janelas rolantes ao longo da trajetória. (f), A direção da etapa Ci traçada ao longo do tempo, com o sombreamento denotando incerteza. (g), Gráfico de ocupação ATOM com tempo de residência (escala de cores). O tamanho do compartimento corresponde ao erro de localização. Regiões notáveis de ocupação estendida, marcados como loops e redemoinhos (i) - (iii), são indicativos de estruturas nanoscópicas persistentes. A região fechada representa uma mancha densa de subdifusão notável. Barras de escala, 100 nm. Crédito: Nature Photonics , doi:10.1038 / s41566-019-0414-6
Taylor et al. verificamos esses dois casos concretos no presente trabalho comparando PNB de diâmetros variados em 48 nm e 20 nm. Os cientistas então conduziram estudos fluorescentes e bioquímicos para sugerir que os GNPs revestidos com EGF ativavam a sinalização do EGFR, muito parecido com os EGFs disponíveis gratuitamente, indicando que o rótulo não prejudica as funções biológicas. Para superar o ruído de fundo relacionado à imagem molecular, os cientistas implementaram um novo algoritmo, que extraiu a função de difusão de ponto iSCAT completa (iSCAT-PSF) diretamente de cada quadro para maior clareza. Uma vez que as técnicas existentes são incapazes de visualizar recursos em alta resolução espacial e temporal, muitos detalhes sobre a atividade intracelular permanecem uma questão de debate. Em resposta, o novo método de Taylor et al. ofereceu uma grande variedade de heterogeneidades dinâmicas em 3-D para lançar luz sobre o movimento da proteína intracelular.
Os cientistas primeiro estudaram quantitativamente a subdifusão na membrana plasmática, considerando um exemplo 2-D da jornada do EGFR na membrana de uma célula HeLa viva. Por esta, eles calcularam o deslocamento quadrático médio (MSD) para toda a trajetória de movimento. Taylor et al. não precisou fazer suposições sobre a natureza da difusão ou sua paisagem geográfica durante o cálculo. Eles mediram a ocorrência de barreiras difrativas biológicas e confinamentos observando o grau de correlação direcional entre duas etapas vetoriais em um intervalo de tempo.

Difusão em um filopódio. Crédito: Nature Photonics , doi:10.1038 / s41566-019-0414-6
Os cientistas então avaliaram a popularidade de cada pixel de trajetória no espaço, introduzindo um mapa de ocupação temporal acumulada (ATOM). Nesta técnica, eles dividiram o plano lateral da trajetória em caixas do tamanho de nanômetros e contaram a ocorrência da partícula em cada caixa. Os resultados indicaram o arranjo de nanoestruturas em loops e redemoinhos dentro de um tempo de vida mínimo de 250 nanossegundos (5000 frames) para retratar potencialmente uma etapa pré-endocítica. No total, as observações simuladas mostraram como a difusão da proteína foi afetada pela subestrutura da célula.
A técnica de microscopia iSCAT permitiu aos cientistas registrar os efeitos por um longo período de tempo, que eles usaram junto com recursos de imagem 3-D para seguir EGFRs em um filopódio. Os filópodes são protusões celulares biologicamente semelhantes a bastonetes contendo feixes de filamentos de actina de até 100 a 300 nm de diâmetro e 100 µm de comprimento. As nanoestruturas podem detectar estímulos mecânicos para quimioatração ou repulsão no microambiente celular, ao mesmo tempo em que fornecem locais para fixação celular. A ligação do ligante e a ativação do EFGR em filópodes ocorreram em baixas concentrações de EGF, seguido por sua associação com filamentos de actina e transporte retrógrado de EFGR para o corpo celular.
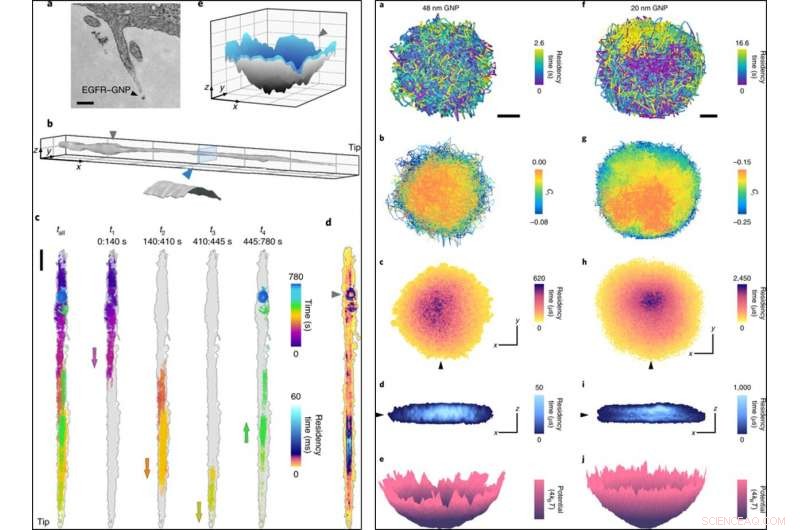
À ESQUERDA:(a) Uma imagem TEM (microscópio eletrônico de transmissão) de um filopódio incluindo um EGFR-GNP. (b), Uma superfície de filopódio reconstruída a partir de 780, 000 pontos de trajetória com um erro de localização de σx, y =2 nm registrado em 1, 000 fps. Inserir, corte transversal que representa uma superfície cilíndrica de diâmetro de 150 nm após levar em conta o tamanho do PNB. (c), Uma trajetória bruta de 13 min (esquerda) dividida em quatro peças subsequentes que revelam a jornada de e para a ponta, com setas marcando a direção do movimento da rede. (d), Um gráfico ATOM de c, corrigido para deriva de filopódio. (e), Uma interpolação de superfície dos 80 s finais. O confinamento em forma de anel na fase final (marcado com um triângulo) é um poço 3D. As barras de escala são 200 nm (a), 1 μm (x, y) e 200 nm (z) (b), 1, 000 nm (c) e 100 nm (x, y) e 50 nm (z) (e). DIREITO:(a), Uma trajetória lateral de uma sonda GNP de 48 nm. Barra de escala, 100 nm. Uma amostragem temporal inferior desse confinamento teria subestimado a extensão do limite. (b), Ci da trajetória (usando um intervalo de tempo de cinco quadros), que mostra a difusão parcialmente impedida com uma propensão para uma difusão mais livre no centro. (c), Um gráfico ATOM de a. (d), Um corte no gráfico 3D-ATOM ao longo da linha do triângulo preto em c mostra que a ocupação favorece uma região mais interna semelhante a um disco. Os eixos denotam 100 nm em c e d. (e), Conversão da ocupação 2D temporal de c em uma distribuição efetiva de energia potencial. (f – j), Equivalente a a – e, mas para uma sonda de GNP de 20 nm. Crédito: Nature Photonics , doi:10.1038 / s41566-019-0414-6
Os cientistas ganharam uma visão sobre os detalhes nanoscópicos da difusão ao longo do filopódio e registraram os dados ao longo de 13 minutos. Eles analisaram a trajetória 3-D para criar a topografia do filopódio usando nanopartículas de ouro como um 'nano rover' e mapearam a topologia de superfície das estruturas celulares para um exame mais profundo. Eles traçaram a trajetória ATOM (mapa de ocupação temporal acumulada) e descobriram que a representação 3-D era consistente com a etapa biológica da invaginação da membrana pré-endocítica.
Técnicas de microscopia de alta velocidade, como iSCAT, são necessárias para obter informações temporais de alta resolução e evitar efeitos de desfoque durante a imagem baseada em localização de nanopartículas. Os cientistas demonstraram esse recurso registrando a difusão confinada em 30, 000 fps (quadros por segundo) com GNPs de 48 nm e 20 nm. Eles seguiram os experimentos com rastreamento 3-D de ultra-alta velocidade de proteínas em 66, 000 fps usando um curto tempo de exposição de 10 µs em um período de tempo de 3,5 segundos. A imagem de microscopia iSCAT rápida forneceu evidências adicionais para revelar as características intrincadas de eventos endocíticos em relação à endocitose mediada por clatrina em células HeLa quando simuladas por baixas concentrações de EGF.
Desta maneira, Taylor et al. observou que a nova técnica poderia registrar fielmente as informações nano-topográficas. Os resultados corresponderam às observações registradas com microscopia eletrônica de transmissão (TEM), sem diferenças significativas na redução do tamanho da sonda de 48 nm a 20 nm, ao mesmo tempo em que fornece novos insights. Os novos insights incluíram detalhes de subdifusão, confinamento nanoscópico, Contornos 3-D de estruturas de filópodes e clatrina em nanoescala. Os cientistas pretendem combinar iSCAT com microscopia de fluorescência de super-resolução in situ para entender as trajetórias das proteínas, vírus e outras entidades biológicas nanoscópicas. Taylor et al. objetivam avançar os métodos de análise de imagem para rastrear GNPs menores que 20 nm no futuro e acreditam que a nova tecnologia e a otimização adicional permitirão que eles entendam especificamente o ciclo de vida dos vírus sem usar um rótulo externo para rastreamento.
© 2019 Science X Network