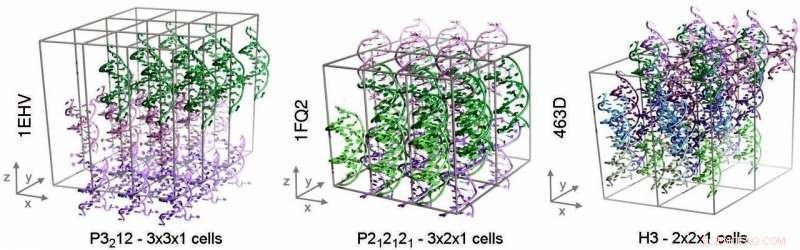
Os três sistemas de cristal examinados neste estudo. Esses sistemas contêm (da esquerda para a direita) 27, 24 e 36 DNA de fita dupla, respectivamente. Crédito:Pablo Dans Puiggròs, IRB Barcelona
Desde o nascimento da biologia estrutural, A cristalografia de raios-X tem sido a técnica mais amplamente usada para determinar a estrutura tridimensional de biomoléculas, os compostos químicos encontrados em organismos vivos. A respeito disso, o conhecimento das interações entre as biomoléculas com seu ambiente cristalino e as forças moleculares que estabilizam os cristais serviriam para otimizar essa técnica.
Um estudo publicado na revista Chem e realizado por pesquisadores do Instituto de Pesquisa em Biomedicina (IRB Barcelona) é o primeiro a alcançar simulações estáveis de cristais de DNA. Essa conquista permitiu aos cientistas explicar a importância dos aditivos químicos usados experimentalmente para se obter condições de cristalização adequadas e cristais estáveis em laboratório.
"O primeiro a se beneficiar deste estudo é a comunidade de biofísicos e físicos / químicos computacionais, que agora têm uma referência e protocolos claros para conseguir simulações estáveis de cristais de DNA, "diz Pablo D. Dans, pesquisador de pós-doutorado no IRB Barcelona.
Liderado por Modesto Orozco, chefe do laboratório de Modelagem Molecular e Bioinformática, o estudo apresenta a descrição atômica mais detalhada das propriedades dos cristais de DNA até o momento.
"A longo prazo, a simulação de vários cristais obtidos nas mais diversas condições experimentais deve permitir-nos antecipar e prever o efeito de um determinado aditivo químico, servindo assim para guiar os cristalógrafos em seus experimentos e reduzindo consideravelmente o custo e o tempo necessário para a obtenção dos cristais, "diz Modesto Orozco, cujo laboratório é referência internacional em simulação e computação bimolecular.