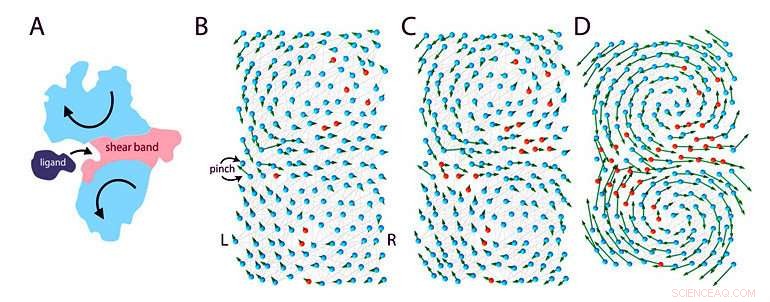
Figura 1:Modelo elástico de uma proteína ligando-se a um ligante. (A) Quando uma proteína se liga a um ligante, ele sofre movimentos em grande escala (setas), que são as assinaturas de proteínas funcionais de dobra. Isso só é possível graças à presença de certas regiões “flexíveis” (“faixa de cisalhamento” rosa) ao longo da proteína que separa as regiões rígidas (azuis) da proteína em dois domínios. (B) - (D) A equipe modelou uma proteína de 200 aminoácidos durante diferentes estágios de evolução:passando de um estado não funcional (B) para um funcional (D). A proteína é modelada como uma rede de mola elástica com dois tipos de aminoácidos, modelado como contas:os aminoácidos rosa são flexíveis e os aminoácidos azuis são rígidos. Os pesquisadores imitam a evolução alterando um aminoácido aleatório de cada vez (mutação) de rosa para azul. Inicialmente, a proteína é principalmente rígida e não funcional. Durante a evolução, aminoácidos flexíveis são adicionados, alguns úteis, alguns não. Hora extra, uma região "flexível" se forma no centro da molécula, tornando a proteína mais flexível para dobrar e se ligar ao ligante. O modelo estimou que uma solução eficiente é alcançada após mil mutações. Crédito:Institute for Basic Science
Uma equipe internacional desenvolveu um modelo que simula a evolução de proteínas. Começando do duro, proteínas não funcionais, o modelo de computador mostra como os componentes da proteína em evolução podem trabalhar juntos para dar origem a máquinas moleculares dinâmicas e eficientes. A flexibilidade permite que as proteínas alterem sua conformação 3D para se ligarem a outras moléculas:essa propriedade é crucial para sua função. Prof. Tsvi Tlusty e Dr. Sandipan Dutta no Center for Soft and Living Matter, dentro do Institute for Basic Science (IBS, Coreia do Sul), em colaboração com o Prof. Albert Libchaber da Rockefeller University e o Prof. Jean-Pierre Eckmann da University of Geneva, imitaram a evolução do gene para obter proteínas que podem dobrar e se ligar a outras moléculas. A compreensão dessa relação é um dos aspectos mais procurados da biologia de proteínas; poderia ajudar a explicar a ação farmacêutica das drogas que se ligam a seus alvos.
A evolução moldou o mundo vivo que vemos ao nosso redor por bilhões de anos. Zilhões de proteínas funcionam harmoniosamente para manter esses processos vitais em andamento. Eles são responsáveis pelo bom funcionamento de qualquer organismo:eles reconhecem outras moléculas (ligantes), ligar-se a eles e convertê-los. Outros têm função de transporte, fornecer estrutura, e suporte às células. Os genes armazenam as informações sobre a produção e o design dessas máquinas moleculares. Contudo, apesar de décadas de pesquisa, traçar o "mapa" que traça o caminho dos genes à função da proteína não é trivial.
De acordo com uma hipótese recente, a função da proteína depende de "juntas flexíveis". Este estudo, publicado em Proceedings of the National Academy of Sciences ( PNAS ), examina a ligação entre função e flexibilidade modelando proteínas como redes elásticas. Neste modelo, as proteínas são feitas de aminoácidos flexíveis (polares) e rígidos (hidrofóbicos) conectados por "molas" moleculares. Se algumas regiões da proteína são flexíveis o suficiente, eles formam um canal "flexível", e toda a máquina molecular pode se dobrar como uma dobradiça. Esse movimento permite que eles se liguem efetivamente a outras moléculas. A ligação entre um ligante e uma proteína rígida ou flexível pode ser pensada como uma bola caindo em uma rocha ou um travesseiro macio. É provável que a bola quique para longe depois de atingir a rocha, mas é mais provável que o travesseiro o aceite. Portanto, a proteína flexível é um aglutinante melhor.
Neste modelo, os genes armazenam os detalhes do projeto da proteína de uma forma binária:os aminoácidos flexíveis são armazenados como zeros e os aminoácidos rígidos como uns. Como resultado, toda a estrutura da proteína pode ser simplificada como um código, como 11110001 ... 111, semelhante à memória digital de um computador. Contudo, nem todos os códigos dão origem a proteínas funcionais, por exemplo, um código com apenas uns:111111… 1111, daria origem a uma proteína totalmente rígida, incapaz de mover-se, e não funcional. Entre todos os códigos possíveis, apenas alguns produzem uma proteína funcional com uma região "flexível" no centro que pode receber o ligante.
O modelo imita a evolução alterando um aminoácido aleatório de cada vez. Durante a evolução, os zeros e uns no gene são invertidos aleatoriamente por um processo chamado mutação. A maioria das mutações não trazem nenhuma diferença, ou levar a proteínas não funcionais, mas algumas mutações raras podem dar origem a uma proteína mais eficiente. Essencialmente, proteínas funcionais e não funcionais são produzidas durante a evolução, mas de acordo com a teoria de Darwin de "sobrevivência do mais apto", apenas as proteínas funcionais são mantidas e as proteínas não funcionais eventualmente morrem.
Qual é a aparência de um código "funcional"? A resposta não é direta. Na verdade, o número de códigos de uma proteína funcional, mesmo uma proteína simples, é enorme, maior do que o tamanho do universo. Contudo, usando técnicas de análise de dados, é possível pesquisar padrões ocultos em todos os códigos funcionais para procurar algumas características unificadoras. Por exemplo, o canal "flexível" na proteína tem características interessantes e peculiares, e uma mutação em uma extremidade do canal tem efeitos de longo alcance que podem afetar fortemente a manutenção de mutações de outros aminoácidos distantes.
"No futuro, pretendemos explorar como aplicar este estudo a proteínas reais, como quinases, "disse o líder do grupo, Tsvi Tlusty, correspondente no estudo. "Além disso, o estudo abre caminhos para investigar a evolução de outras funções de proteínas, como o reconhecimento molecular. Usando bancos de dados enormes, que foram desenvolvidos ao longo de anos de pesquisa, pode provavelmente descobrir alguns fenômenos subjacentes na evolução das proteínas. "