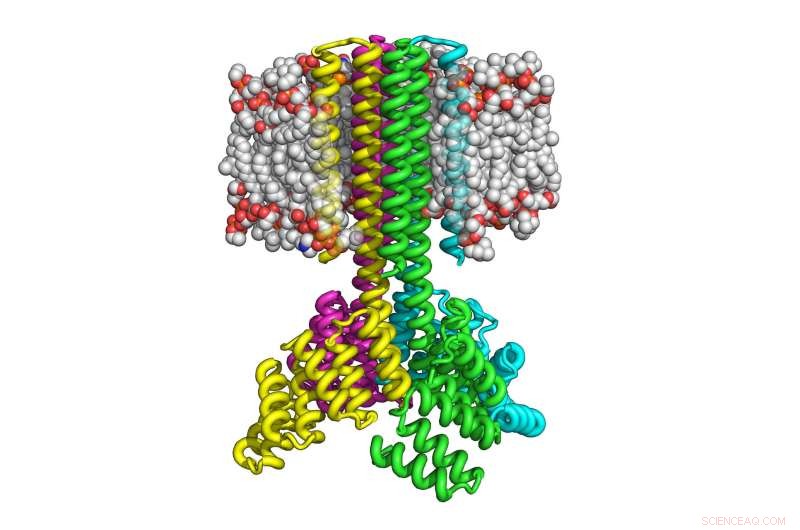
Esta ilustração mostra como quatro cópias da proteína transmembrana projetada por computador se reuniram para formar um tetrâmero em forma de foguete com uma ampla base citoplasmática que afunila em oito hélices transmembrana e que se orienta corretamente na membrana. Crédito:University of Washington Institute for Protein Design
Agora é possível criar complexos, proteínas transmembrana personalizadas do zero, cientistas relatam esta semana. O avanço, liderado por engenheiros moleculares do Instituto de Design de Proteínas da Universidade de Washington, permitirá aos pesquisadores criar proteínas transmembrana não encontradas na natureza para realizar tarefas específicas.
No mundo dos vivos, proteínas transmembrana são encontradas embutidas na membrana de todas as células e organelas celulares. Eles são essenciais para que funcionem normalmente. Por exemplo, muitas proteínas transmembrana que ocorrem naturalmente atuam como portas de entrada para o movimento de substâncias específicas através de uma membrana biológica. Algumas proteínas transmembrana recebem ou transmitem sinais celulares. Por causa de tais funções, muitos medicamentos são projetados para atingir as proteínas transmembrana e alterar sua função.
"Nossos resultados abrem o caminho para o projeto de proteínas de membrana multispan que podem imitar proteínas encontradas na natureza ou ter uma estrutura inteiramente nova, função e usos, "disse David Baker, um professor de bioquímica da University of Washington School of Medicine e diretor do UW Institute of Protein Design, que liderou o projeto. A pesquisa foi publicada na edição de 1º de março da revista. Ciência . Peilong Lu, um colega sênior no laboratório Baker, é o autor principal do artigo.
Mas entender como as proteínas transmembrana são reunidas e como funcionam tem se mostrado um desafio. Porque eles agem enquanto embutidos na membrana celular, proteínas transmembrana têm se mostrado mais difíceis de estudar do que proteínas que atuam na solução aquosa que compõe o citoplasma das células ou no fluido extracelular.
No novo estudo, Lu e seus colegas de trabalho usaram um programa de computador, desenvolvido no laboratório Baker e chamado Rosetta, que pode prever a estrutura em que uma proteína se dobrará após ter sido sintetizada. A arquitetura de uma proteína é crucial porque a estrutura de uma proteína determina sua função.
A forma de uma proteína se forma a partir de interações complexas entre os aminoácidos que compõem a cadeia protéica e entre os aminoácidos e o ambiente circundante. Em última análise, a proteína assume a forma que melhor equilibra todos esses fatores para que a proteína atinja o estado de energia mais baixo possível.
O programa Rosetta usado por Lu e seus colegas pode prever a estrutura de uma proteína levando em consideração essas interações e calculando o estado geral de energia mais baixo. Não é incomum que o programa crie dezenas de milhares de estruturas modelo para uma sequência de aminoácidos e, em seguida, identifique aquelas com o estado de energia mais baixo. Os modelos resultantes demonstraram representar com precisão a estrutura que a sequência provavelmente assumirá na natureza.
Determinar a estrutura das proteínas transmembrana é difícil porque porções das proteínas transmembrana devem passar pelo interior da membrana, que é feito de gorduras oleosas chamadas lipídios.
Em fluidos aquosos, resíduos de aminoácidos que têm cadeias laterais polares - componentes que podem ter uma carga sob certas condições fisiológicas ou que participam de ligações de hidrogênio - tendem a estar localizados na superfície da proteína, onde podem interagir com a água, que tem cargas laterais negativas e positivas em sua molécula. Como resultado, resíduos polares em proteínas são chamados de hidrofílicos, ou "amante da água".
Resíduos não polares, por outro lado, tendem a ser encontrados empacotados dentro do núcleo da proteína, longe do fluido aquoso polar. Esses resíduos são chamados de hidrofóbicos ou "temerosos de água". Como resultado, a interação entre os resíduos da proteína que amam a água e temem a água e os fluidos aquosos circundantes ajuda a conduzir o dobramento da proteína e estabiliza a estrutura final da proteína.
Em membranas, Contudo, o dobramento de proteínas é mais complicado porque o interior de lipídios da membrana não é polar, isso é, não tem separação de cargas elétricas. Isso significa que para ser estável a proteína deve ser não polar, resíduos temerosos de água em sua superfície, e empacote seu polar, resíduos que amam a água dentro. Em seguida, ele deve encontrar uma maneira de estabilizar sua estrutura criando ligações entre os resíduos hidrofílicos dentro de seu núcleo.
A chave para resolver o problema, diz o Lu, era aplicar um método desenvolvido pelo laboratório Baker para projetar proteínas para que os pólos, resíduos hidrofílicos se encaixam de tal forma que o suficiente formaria interações polar-polares que podem unir a proteína por dentro.
"Juntar essas 'redes de ligações de hidrogênio enterradas' foi como montar um quebra-cabeça, "Baker disse.
Com esta abordagem, Lu e seus colegas foram capazes de fabricar as proteínas transmembrana projetadas dentro de bactérias e células de mamíferos usando até 215 aminoácidos. As proteínas resultantes mostraram-se altamente estáveis termicamente e capazes de se orientar corretamente na membrana. Como proteínas transmembrana de ocorrência natural, as proteínas são multipass, o que significa que eles atravessam a membrana várias vezes, e montar em complexos multi-proteínas estáveis, como dímeros, trímeros e tetrâmeros.
"Nós mostramos que agora é possível projetar complexos com precisão, proteínas transmembrana multipass que podem ser expressas em células. Isso possibilitará aos pesquisadores projetar proteínas transmembrana com estruturas e funções inteiramente novas, "disse o Lu.