Como diferentes células cancerígenas respondem a nanopartículas que liberam drogas
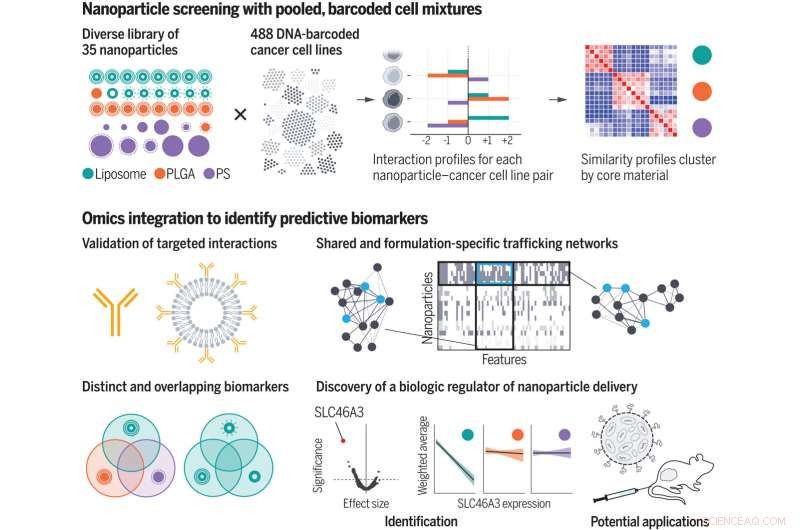
Usando uma biblioteca de nanopartículas com curadoria, os pesquisadores examinaram os perfis de interação nanopartícula-célula de centenas de células cancerígenas simultaneamente. Ao incorporar a anotação ômica, eles identificaram características biológicas, ou biomarcadores, que mediam a entrega de nanopartículas às células. Eles geraram redes de tráfico e descobriram um regulador biológico da entrega de nanopartículas à base de lipídios. PLGA, polilactido-co-glicolido; PS, poliestireno. Crédito:Natalie Boehnke et al, Ciência (2022). DOI:10.1126/science.abm5551
O uso de nanopartículas para administrar medicamentos contra o câncer oferece uma maneira de atingir tumores com grandes doses de medicamentos, evitando os efeitos colaterais prejudiciais que geralmente acompanham a quimioterapia. No entanto, até agora, apenas um punhado de medicamentos contra o câncer à base de nanopartículas foram aprovados pela FDA.
Um novo estudo do MIT e do Broad Institute of MIT e pesquisadores de Harvard pode ajudar a superar alguns dos obstáculos ao desenvolvimento de drogas baseadas em nanopartículas. A análise da equipe das interações entre 35 tipos diferentes de nanopartículas e quase 500 tipos de células cancerígenas revelou milhares de características biológicas que influenciam se essas células absorvem diferentes tipos de nanopartículas.
As descobertas podem ajudar os pesquisadores a adaptar melhor suas partículas de entrega de medicamentos a tipos específicos de câncer ou projetar novas partículas que aproveitem as características biológicas de tipos específicos de células cancerígenas.
"Estamos entusiasmados com nossas descobertas porque é realmente apenas o começo - podemos usar essa abordagem para mapear quais tipos de nanopartículas são melhores para atingir certos tipos de células, de câncer a células imunes e outros tipos de células de órgãos saudáveis e doentes. Estamos aprendendo como a química da superfície e outras propriedades do material desempenham um papel no direcionamento”, diz Paula Hammond, professora do Instituto MIT, chefe do Departamento de Engenharia Química e membro do Koch Institute for Integrative Cancer Research do MIT.
Hammond é o autor sênior do novo estudo, que aparece na
Science . Os principais autores do artigo são Natalie Boehnke, pós-doutoranda do MIT que em breve se juntará ao corpo docente da Universidade de Minnesota, e Joelle Straehla, investigadora clínica de Charles W. e Jennifer C. Johnson no Koch Institute, instrutora da Harvard Medical School, e um oncologista pediátrico no Dana-Farber Cancer Institute.
Interações célula-partícula O laboratório de Hammond desenvolveu anteriormente muitos tipos de nanopartículas que podem ser usadas para fornecer drogas às células. Estudos em seu laboratório e outros mostraram que diferentes tipos de células cancerígenas geralmente respondem de maneira diferente às mesmas nanopartículas. Boehnke, que estudava câncer de ovário quando ingressou no laboratório de Hammond, e Straehla, que estudava câncer no cérebro, também notaram esse fenômeno em seus estudos.
Os pesquisadores levantaram a hipótese de que as diferenças biológicas entre as células poderiam estar conduzindo a variação em suas respostas. Para descobrir quais poderiam ser essas diferenças, eles decidiram realizar um estudo em larga escala no qual pudessem observar um grande número de células diferentes interagindo com muitos tipos de nanopartículas.
Straehla tinha aprendido recentemente sobre a plataforma PRISM do Broad Institute, que foi projetada para permitir que os pesquisadores rastreiem rapidamente milhares de medicamentos em centenas de tipos diferentes de câncer ao mesmo tempo. Com a colaboração instrumental de Angela Koehler, professora associada de engenharia biológica do MIT, a equipe decidiu tentar adaptar essa plataforma para rastrear interações célula-nanopartículas em vez de interações célula-droga.
"Usando essa abordagem, podemos começar a pensar se há algo na assinatura genotípica de uma célula que prevê quantas nanopartículas ela vai absorver", diz Boehnke.
Para a triagem, os pesquisadores usaram 488 linhagens de células cancerígenas de 22 tecidos de origem diferentes. Cada tipo de célula é "codificado de barras" com uma sequência de DNA única que permite aos pesquisadores identificar as células mais tarde. Para cada tipo de célula, extensos conjuntos de dados também estão disponíveis em seus perfis de expressão gênica e outras características biológicas.
No lado das nanopartículas, os pesquisadores criaram 35 partículas, cada uma com um núcleo composto por lipossomas (partículas feitas de muitas moléculas gordurosas chamadas lipídios), um polímero conhecido como PLGA ou outro polímero chamado poliestireno. Os pesquisadores também revestiram as partículas com diferentes tipos de moléculas protetoras ou de direcionamento, incluindo polímeros como polietilenoglicol, anticorpos e polissacarídeos. Isso permitiu que eles estudassem a influência da composição do núcleo e da química da superfície das partículas.
Trabalhando com cientistas do Broad Institute, incluindo Jennifer Roth, diretora do laboratório PRISM, os pesquisadores expuseram conjuntos de centenas de células diferentes a uma das 35 nanopartículas diferentes. Cada nanopartícula tinha uma etiqueta fluorescente, para que os pesquisadores pudessem usar uma técnica de classificação de células para separar as células com base na quantidade de fluorescência que elas emitiam após uma exposição de quatro ou 24 horas.
Com base nessas medições, cada linha celular recebeu uma pontuação que representa sua afinidade para cada nanopartícula. Os pesquisadores então usaram algoritmos de aprendizado de máquina para analisar essas pontuações junto com todos os outros dados biológicos disponíveis para cada linhagem celular.
Essa análise rendeu milhares de recursos, ou biomarcadores, associados à afinidade por diferentes tipos de nanopartículas. Muitos desses marcadores eram genes que codificam a maquinaria celular necessária para ligar partículas, trazê-las para uma célula ou processá-las. Alguns desses genes já eram conhecidos por estarem envolvidos no tráfico de nanopartículas, mas muitos outros eram novos.
"Encontramos alguns marcadores que esperávamos, e também encontramos muito mais que realmente não foi explorado. Esperamos que outras pessoas possam usar esse conjunto de dados para ajudar a expandir sua visão de como as nanopartículas e as células interagem", diz Straehla.
Captação de partículas Os pesquisadores escolheram um dos biomarcadores que identificaram, uma proteína chamada SLC46A3, para um estudo mais aprofundado. A tela PRISM mostrou que altos níveis dessa proteína se correlacionavam com uma absorção muito baixa de nanopartículas à base de lipídios. Quando os pesquisadores testaram essas partículas em modelos de melanoma em camundongos, eles encontraram a mesma correlação. As descobertas sugerem que esse biomarcador pode ser usado para ajudar os médicos a identificar pacientes cujos tumores são mais propensos a responder a terapias baseadas em nanopartículas.
Agora, os pesquisadores estão tentando descobrir o mecanismo de como SLC46A3 regula a absorção de nanopartículas. Se eles pudessem descobrir novas maneiras de diminuir os níveis celulares dessa proteína, isso poderia ajudar a tornar os tumores mais suscetíveis a drogas transportadas por nanopartículas lipídicas. Os pesquisadores também estão trabalhando para explorar ainda mais alguns dos outros biomarcadores que encontraram.
Essa abordagem de triagem também pode ser usada para investigar muitos outros tipos de nanopartículas que os pesquisadores não analisaram neste estudo.
“O céu é o limite em termos de outros biomarcadores não descobertos que simplesmente não capturamos porque não os examinamos”, diz Boehnke. “Espero que seja uma inspiração para outros começarem a olhar para seus sistemas de nanopartículas de maneira semelhante”.
+ Explorar mais Engenheiros desenvolvem nanopartículas que atravessam a barreira hematoencefálica
Esta história foi republicada como cortesia do MIT News (web.mit.edu/newsoffice/), um site popular que cobre notícias sobre pesquisa, inovação e ensino do MIT.
