
SINGLE usa imagens TEM in situ de nanocristais de platina girando livremente em uma célula líquida de grafeno para determinar as estruturas 3-D de nanopartículas coloidais individuais. Crédito:Berkeley Lab
Assim como as proteínas são um dos blocos básicos de construção da biologia, nanopartículas podem servir como blocos de construção básicos para materiais de próxima geração. Em consonância com este paralelo entre biologia e nanotecnologia, uma técnica comprovada para determinar as estruturas tridimensionais de proteínas individuais foi adaptada para determinar as estruturas 3D de nanopartículas individuais em solução.
Uma equipe multi-institucional de pesquisadores liderada pelo Laboratório Nacional Lawrence Berkeley do Departamento de Energia dos EUA (DOE) (Berkeley Lab), desenvolveu uma nova técnica chamada "SINGLE", que fornece as primeiras imagens em escala atômica de nanopartículas coloidais. SOLTEIRO, que significa Identificação de Estrutura 3D de Nanopartículas por Microscopia Eletrônica de Célula Líquida de Grafeno, foi usado para reconstruir separadamente as estruturas 3D de duas nanopartículas de platina individuais em solução.
"Compreender os detalhes estruturais das nanopartículas coloidais é necessário para construir uma ponte sobre o nosso conhecimento sobre a sua síntese, mecanismos de crescimento, e propriedades físicas para facilitar sua aplicação à energia renovável, catálise e muitos outros campos, "diz o diretor do Berkeley Lab e renomada autoridade em nanociência, Paul Alivisatos, quem liderou esta pesquisa. "Considerando que a maioria dos estudos estruturais de nanopartículas coloidais são realizados no vácuo após a conclusão do crescimento do cristal, nosso método SINGLE nos permite determinar sua estrutura 3D em uma solução, um passo importante para melhorar o design de nanopartículas para aplicações de catálise e pesquisa de energia. "
Alivisatos, que também detém a Cátedra Distinta da Samsung em Nanociência e Nanotecnologia na Universidade da Califórnia em Berkeley, e dirige o Instituto de Nanociências de Energia Kavli em Berkeley (Kavli ENSI), é o autor correspondente de um artigo que detalha essa pesquisa na revista Ciência . O artigo é intitulado "Estrutura 3D de Nanocristais Individuais em Solução por Microscopia Eletrônica". Os co-autores principais são Jungwon Park da Harvard University, Hans Elmlund, da Universidade Monash da Austrália, e e Peter Ercius do Laboratório de Berkeley. Outros co-autores são Jong Min Yuk, David Limmer, Qian Chen, Kwanpyo Kim, Sang Hoon Han, David Weitz e Alex Zettl.
Nanopartículas coloidais são aglomerados de centenas a milhares de átomos suspensos em uma solução cujas propriedades químicas e físicas coletivas são determinadas pelo tamanho e forma das nanopartículas individuais. As técnicas de imagem que são usadas rotineiramente para analisar a estrutura 3D de cristais individuais em um material não podem ser aplicadas a nanomateriais suspensos porque as partículas individuais em uma solução não são estáticas. A funcionalidade das proteínas também é determinada por seu tamanho e forma, e os cientistas que queriam criar imagens de estruturas de proteínas em 3D enfrentaram um problema semelhante. O problema de imagem da proteína foi resolvido por uma técnica chamada "microscopia crioeletrônica de partícula única, "em que dezenas de milhares de imagens de microscópio eletrônico de transmissão (TEM) 2D de cópias idênticas de uma proteína individual ou complexo de proteínas congeladas em orientações aleatórias são registradas e depois combinadas computacionalmente em reconstruções 3D de alta resolução. Alivisatos e seus colegas utilizaram este conceito para criar sua técnica ÚNICA.
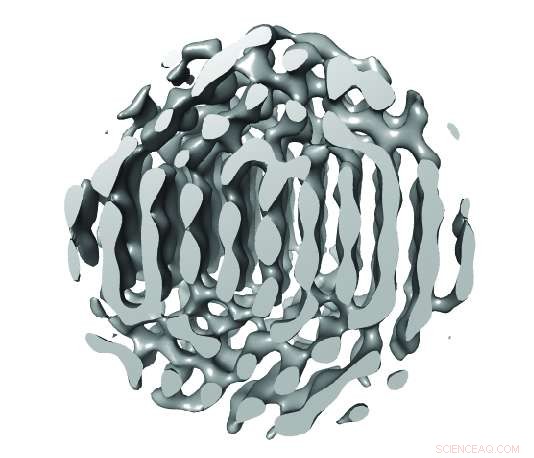
Uma placa através da reconstrução 3-D da partícula 1 ao longo do plano vertical com as posições atômicas provisórias indicadas. Repetições ABC de {111} aviões são visíveis. Crédito:ARC Center of Excellence in Advanced Molecular Imaging
"Na ciência dos materiais, não podemos assumir que as nanopartículas em uma solução são todas idênticas, então precisamos desenvolver uma abordagem híbrida para reconstruir as estruturas 3D de nanopartículas individuais, "diz o co-autor principal do Ciência papel Peter Ercius, um cientista da equipe do National Center for Electron Microscopy (NCEM) na Molecular Foundry, um DOE Office of Science User Facility.
"SINGLE representa uma combinação de três avanços tecnológicos de imagens TEM na ciência biológica e de materiais, "Ercius diz." Esses três avanços são o desenvolvimento de uma célula líquida de grafeno que permite a imagem TEM de nanopartículas girando livremente em solução, detectores de elétrons diretos que podem produzir filmes com resolução de tempo quadro a quadro em milissegundos dos nanocristais rotativos, e uma teoria para a reconstrução 3D de partícula única ab initio. "
A célula líquida de grafeno (GLC) que ajudou a tornar este estudo possível também foi desenvolvida no Berkeley Lab sob a liderança da Alivisatos. A imagem TEM usa um feixe de elétrons em vez de luz para iluminação e ampliação, mas só pode ser usada em alto vácuo porque as moléculas do ar interrompem o feixe de elétrons. Uma vez que os líquidos evaporam em alto vácuo, as amostras em soluções devem ser hermeticamente seladas em recipientes sólidos especiais - chamados de células - com uma janela de visualização muito fina antes de serem fotografadas com TEM. No passado, as células líquidas apresentavam janelas de visualização baseadas em silício, cuja espessura limitava a resolução e perturbava o estado natural dos materiais da amostra. O GLC desenvolvido no laboratório de Berkeley apresenta uma janela de visualização feita de uma folha de grafeno com apenas um único átomo de espessura.
"O GLC nos fornece uma cobertura ultrafina de nossas nanopartículas, enquanto mantém as condições do líquido no vácuo TEM, "Diz Ercius." Como a superfície de grafeno do GLC é inerte, ele não adsorve ou perturba o estado natural de nossas nanopartículas. "
Trabalhando na EQUIPE I do NCEM, o microscópio eletrônico mais poderoso do mundo, Ercius, Alivisatos e seus colegas foram capazes de obter imagens in situ dos movimentos translacionais e de rotação de nanopartículas individuais de platina com menos de dois nanômetros de diâmetro. Nanopartículas de platina foram escolhidas por causa de sua alta força de espalhamento de elétrons e porque sua estrutura atômica detalhada é importante para a catálise.
"Nossos estudos anteriores de GLC de nanocristais de platina mostraram que eles crescem por agregação, resultando em estruturas complexas que não são possíveis de determinar por qualquer método desenvolvido anteriormente, "Diz Ercius." Como a SINGLE deriva suas estruturas 3D de imagens de nanopartículas individuais girando livremente em solução, permite a análise de populações heterogêneas de nanopartículas potencialmente desordenadas que são sintetizadas em solução, proporcionando assim um meio para compreender a estrutura e estabilidade dos defeitos em nanoescala. "
A próxima etapa do SINGLE é recuperar um mapa de densidade de resolução atômica 3D completo de nanopartículas coloidais usando uma câmera mais avançada instalada no TEAM I que pode fornecer 400 quadros por segundo e melhor qualidade de imagem.
“Planejamos criar imagens de defeitos em nanopartículas feitas de diferentes materiais, partículas da casca do núcleo, e também ligas feitas de duas espécies atômicas diferentes, "Ercius diz.