Design renovado pode levar computadores biológicos poderosos do tubo de ensaio para a célula
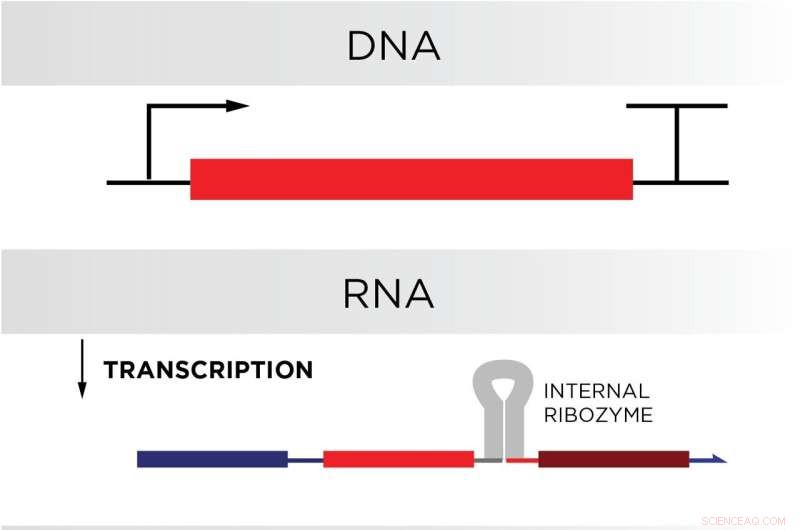
Os pesquisadores do NIST pretendem transformar a célula em uma fábrica de computadores biológicos, projetando e inserindo DNA no genoma de uma célula. As proteínas celulares produziriam RNA com base no DNA através da transcrição. A fita de RNA então se dobraria, ligando-se a si mesma e se dividiria em duas, graças a uma sequência especial de RNA de autoclivagem chamada ribozima. A estrutura resultante, uma porta de circuito de RNA, só seria desfeita e desencadearia mais reações químicas sob certas condições. Crédito:N. Hanacek/NIST
Pequenos computadores biológicos feitos de DNA podem revolucionar a maneira como diagnosticamos e tratamos uma série de doenças, uma vez que a tecnologia esteja totalmente desenvolvida. No entanto, um grande obstáculo para esses dispositivos baseados em DNA, que podem operar tanto em células quanto em soluções líquidas, tem sido sua curta duração. Apenas um uso e os computadores são gastos.
Agora, pesquisadores do Instituto Nacional de Padrões e Tecnologia (NIST) podem ter desenvolvido computadores biológicos de longa duração que poderiam persistir dentro das células. Em artigo publicado na revista
Science Advances , os autores renunciam à abordagem tradicional baseada em DNA, optando por usar o RNA de ácido nucleico para construir computadores. Os resultados demonstram que os circuitos de RNA são tão confiáveis e versáteis quanto seus equivalentes baseados em DNA. Além disso, as células vivas podem ser capazes de criar esses circuitos de RNA continuamente, algo que não é prontamente possível com circuitos de DNA, posicionando ainda mais o RNA como um candidato promissor para computadores biológicos poderosos e duradouros.
Assim como o computador ou dispositivo inteligente em que você provavelmente está lendo isso, os computadores biológicos podem ser programados para realizar diferentes tipos de tarefas.
“A diferença é que, em vez de codificar com uns e zeros, você escreve sequências de A, T, C e G, que são as quatro bases químicas que compõem o DNA”, disse Samuel Schaffter, pesquisador de pós-doutorado do NIST e principal autor do estudo. .
Ao montar uma sequência específica de bases em uma fita de ácido nucleico, os pesquisadores podem ditar a que ela se liga. Uma fita pode ser projetada para se ligar a bits específicos de DNA, RNA ou algumas proteínas associadas a uma doença e, em seguida, desencadear reações químicas com outras fitas no mesmo circuito para processar informações químicas e, eventualmente, produzir algum tipo de saída útil.
Essa saída pode ser um sinal detectável que pode ajudar no diagnóstico médico ou pode ser uma droga terapêutica para tratar uma doença.
No entanto, o DNA não é o material mais resistente e pode se desfazer rapidamente em certas condições. As células podem ser ambientes hostis, pois geralmente contêm proteínas que destroem os ácidos nucleicos. E mesmo que as sequências de DNA permaneçam por tempo suficiente para detectar seu alvo, as ligações químicas que elas formam as tornam inúteis depois.
"Eles não podem fazer coisas como monitorar continuamente os padrões de expressão gênica. Eles são um uso, o que significa que eles apenas fornecem um instantâneo", disse Schaffter.
Sendo um ácido nucleico também, o RNA compartilha muitos dos problemas do DNA quando se trata de ser um bloco de construção de computador biológico. É suscetível à rápida degradação e, depois que uma fita se liga quimicamente a uma molécula alvo, essa fita é finalizada. Mas, ao contrário do DNA, o RNA pode ser um recurso renovável nas condições certas. Para aproveitar essa vantagem, Schaffter e seus colegas primeiro precisavam mostrar que os circuitos de RNA, que as células teoricamente seriam capazes de produzir, poderiam funcionar tão bem quanto o tipo baseado em DNA.
A vantagem do RNA sobre o DNA decorre de um processo celular natural chamado transcrição, em que as proteínas produzem RNA continuamente usando o DNA de uma célula como modelo. Se o DNA no genoma de uma célula codificasse os componentes do circuito em um computador biológico, então a célula produziria os componentes do computador continuamente.
No processo de computação biológica, fitas simples de ácidos nucleicos em um circuito podem facilmente acabar ligadas a outras fitas no mesmo circuito, um efeito indesejado que impede que os componentes do circuito se liguem aos seus alvos pretendidos. O design desses circuitos geralmente significa que diferentes componentes serão ajustes naturais um para o outro.
Para evitar ligações indesejadas, as sequências de DNA que fazem parte de computadores conhecidos como circuitos de deslocamento de fita geralmente são sintetizadas (em máquinas em vez de células) separadamente e em uma forma de fita dupla. Com cada base química em cada fita ligada a uma base na outra, essa fita dupla atua como um portão trancado que só seria desbloqueado se a sequência-alvo aparecesse e tomasse o lugar de uma das fitas.
Schaffter e Elizabeth Strychalski, líder do Grupo de Engenharia Celular do NIST e coautora do estudo, procuraram imitar essa função de "portão bloqueado" em seu circuito de RNA, tendo em mente que, em última análise, as células teriam que produzir esses portões bloqueados. Para preparar as células para o sucesso, os pesquisadores escreveram as sequências para que uma metade das fitas pudesse se ligar à outra metade. Ligando-se dessa maneira, as sequências de RNA se dobrariam sobre si mesmas como um pão de cachorro-quente, garantindo que estivessem em um estado bloqueado.
Mas para funcionar corretamente, os portões precisariam ser dois fios quimicamente ligados, mas distintos, mais parecidos com um pão de hambúrguer ou sanduíche do que um pão de cachorro-quente. A equipe obteve o design de fita dupla em seus portões codificando em um trecho de RNA chamado ribozima perto do ponto de dobramento dos portões. Essa ribozima em particular – retirada do genoma de um vírus da hepatite – se separaria depois que a fita de RNA em que foi incorporada se dobrasse, criando duas fitas separadas.
Os autores testaram se seus circuitos poderiam realizar operações lógicas básicas, como apenas desbloquear seus portões em cenários específicos, como se uma das duas sequências de RNA específicas estivesse presente ou apenas se ambas estivessem ao mesmo tempo. Eles também construíram e examinaram circuitos feitos de várias portas que executavam diferentes operações lógicas em série. Somente quando esses circuitos encontrassem a combinação certa de sequências, seus portões se abririam um a um como dominós.
Os experimentos envolveram a exposição de diferentes circuitos a pedaços de RNA - alguns dos quais os circuitos foram projetados para se conectar - e a medição da saída dos circuitos. Nesse caso, a saída no final de cada circuito era uma molécula repórter fluorescente que acenderia assim que o portão final fosse desbloqueado.
Os pesquisadores também rastrearam a taxa na qual os portões foram desbloqueados à medida que os circuitos processavam as entradas e compararam suas medições com as previsões dos modelos de computador.
"Para mim, eles precisavam funcionar em um tubo de ensaio de forma tão preditiva quanto a computação de DNA. O legal dos circuitos de DNA é que, na maioria das vezes, você pode simplesmente escrever uma sequência em um pedaço de papel e funcionará da maneira você quer", disse Schaffter. "A principal coisa aqui é que descobrimos que os circuitos de RNA eram muito previsíveis e programáveis, muito mais do que eu pensava que seriam, na verdade."
As semelhanças de desempenho entre os circuitos de DNA e RNA podem indicar que pode ser benéfico mudar para o último, já que o RNA pode ser transcrito para reabastecer os componentes de um circuito. E muitos circuitos de DNA existentes que os pesquisadores já desenvolveram para realizar várias tarefas poderiam teoricamente ser trocados por versões de RNA e se comportar da mesma maneira. Para ter certeza, porém, os autores do estudo precisam impulsionar a tecnologia ainda mais.
Neste estudo, os autores demonstraram que os circuitos transcritíveis funcionam, mas ainda não os produziram usando a maquinaria celular real da transcrição. Em vez disso, as máquinas sintetizaram os ácidos nucleicos por meio de um processo semelhante ao usado para produzir DNA para pesquisa. Dar o próximo passo exigiria a inserção de DNA no genoma de um organismo, onde serviria como um modelo para os componentes do circuito de RNA.
"Estamos interessados em colocá-los em bactérias em seguida. Queremos saber:podemos empacotar projetos de circuitos em material genético usando nossa estratégia? Podemos obter o mesmo tipo de desempenho e comportamento quando os circuitos estão dentro das células?" disse Schaffer. "Temos potencial para isso."
+ Explorar mais Cientistas desenvolvem novos circuitos lógicos de DNA
