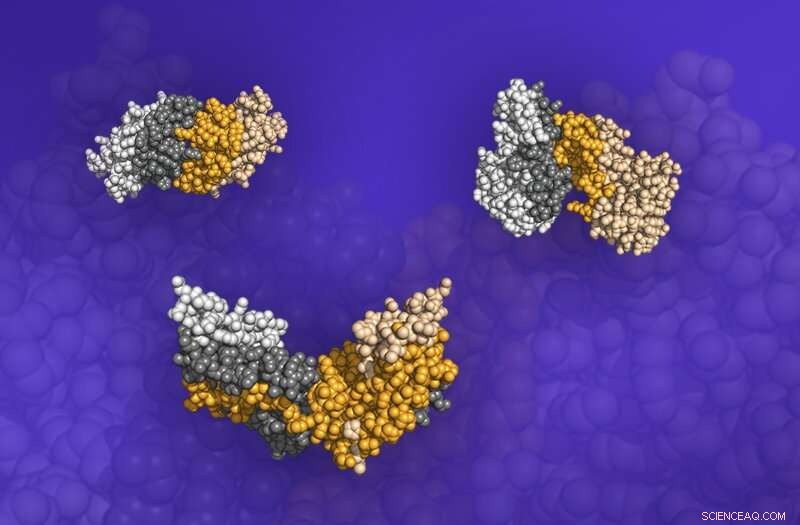
Três dímeros, estruturas de proteínas que consistem em duas proteínas ligadas, do banco de dados Dockground. As interfaces nas quais as proteínas se encontram são mostradas como regiões escurecidas. Crédito:ORNL
Pode algo tão simples como a forma determinar totalmente se as proteínas vão se ligar ou não? Os cientistas estão contratando supercomputadores para descobrir.
Uma equipe liderada por Sharon Glotzer, distinto professor e chefe do departamento de engenharia química da Universidade de Michigan (UM), usou o supercomputador Summit de 200 petaflop no Laboratório Nacional de Oak Ridge (ORNL) do Departamento de Energia dos EUA (DOE) para modelar interações de chave e fechadura entre proteínas para estudar seus comportamentos de ligação. Os resultados, publicado em Matéria Macia, revelou que algumas proteínas fazem, na verdade, vincular com base apenas na forma.
"Demonstramos que algo tão simples como a forma é capaz de prever interações de proteínas que às vezes são muito complexas, "disse Jens Glaser, cientista da computação no grupo de Computação Avançada para Química e Materiais do Oak Ridge Leadership Computing Facility (OLCF). "Esta primeira demonstração nos levou a acreditar que a forma tem sido um ingrediente pouco apreciado em muitos processos de montagem de proteínas."
Os resultados podem ter inúmeras aplicações na pesquisa biológica. Por exemplo, a abordagem pode ser usada para rastrear drogas para doenças ou fornecer aos cientistas informações sobre como usar proteínas como blocos de construção para projetar novos materiais biológicos.
"Este estudo emocionante demonstra o poder da complementaridade da forma na previsão de interfaces proteína-proteína, "disse a Dra. Stephanie McElhinny, gerente de programa do Laboratório de Pesquisa do Exército do Comando de Desenvolvimento de Capacidades de Combate do Exército dos EUA, referindo-se à relação espacial favorável entre duas proteínas de formato compatível. "Os modelos computacionais que prevêem com precisão essas interfaces darão suporte ao projeto futuro de materiais avançados à base de proteínas com propriedades ativas e responsivas, como plásticos baseados em proteínas que captam luz, que podem funcionar como uma folha artificial para geração de energia. "
Supercomputadores revelam que a forma é a chave em algumas proteínas
Para que as proteínas se liguem umas às outras com sucesso, um deles atua como um ligante, uma molécula que se liga a uma proteína alvo, e um deles atua como receptor, a molécula que recebe o ligante. Este processo envolve interações químicas complexas, em que as moléculas compartilham ligações e mudam suas configurações após a ligação.
A equipe de Glotzer queria ver se eles poderiam prever essa ligação molecular com base apenas na forma, ignorando as interações entre as proteínas. De um banco de dados de mais de 6, 000 pares de proteínas, a equipe testou 46 pares que se ligam um ao outro e simulou sua montagem na Summit. A equipe realizou as simulações no programa INCITE (Impacto Computacional Inovador e Novo em Teoria e Experimento).
Como várias bolas de tênis sendo lançadas em um único alvo, as simulações modelaram vários ligantes sendo lançados em um único, receptor alvo fixo. Dos 46 pares testados, eles encontraram 6 pares com bom desempenho - mais de 50 por cento do tempo eles montaram com sucesso com base puramente em suas formas complementares.
"Observamos as interfaces em que as proteínas se ligam para ver como elas eram semelhantes às interfaces da vida real, e então determinamos o corte para ver quantos pares eram bons preditores das interfaces reais, "disse Fengyi Gao, Ph.D. candidato na UM. "Descobrimos que 13 por cento desses pares de proteínas podem se ligar com base apenas na forma."
A equipe, então, construiu um modelo de aprendizado de máquina que poderia determinar quais proteínas são capazes de montar com base exclusivamente na forma. Combinar seu modelo inicial com tais ferramentas de aprendizado de máquina os ajudará a entender quais informações são necessárias para pares de proteínas que não podem ser montados com base apenas na complementaridade da forma.
Executando proteínas em paralelo
Para modelar vários processos de ligação reversíveis de 46 pares de proteínas sob diferentes parâmetros, eles precisavam de dois dias de tempo computacional e mais de 3, 000 GPUs - uma quantidade que apenas um supercomputador como o OLCF's Summit poderia fornecer. O OLCF é um DOE Office of Science User Facility no ORNL.
Como parte do código computacional HOOMD-blue que foi usado para executar as simulações, Glaser, que anteriormente era um cientista assistente de pesquisa no grupo de Glotzer na UM, desenvolveu um algoritmo que simulava as proteínas na presença de muitas partículas pequenas. Mas Glaser encontrou uma maneira de modelar apenas o movimento das proteínas nas quais a equipe estava interessada, evitando cálculos desnecessários e caros para as moléculas de solvente ao seu redor.
"Eu executei o código em paralelo para que muitos parâmetros diferentes, iterações do mesmo sistema, e diferentes proteínas podem ser distribuídas pelas GPUs, "Glaser disse." Isso nos permitiu usar facilmente os recursos de computação paralela da Summit. "
Usando Summit, a equipe capturou seis pares de proteínas que se ligam com base apenas na complementaridade da forma, com um deles atingindo a vinculação em mais de 94% das vezes.
"Foi bastante surpreendente para nós que um modelo tão simplificado pudesse selecionar corretamente apenas aquela pose que eles assumem entre as muitas centenas ou mais poses que competem, "Esperávamos que muito mais fosse necessário para reproduzir a pose real de ligação para esses pares de proteínas", disse Glaser.
Os modelos podem ajudar na triagem de drogas
A equipe planeja estudar mais proteínas que também podem se ligar com base na forma - ou formar estruturas de ordem ainda mais elevada. O estudo atual da equipe explorou apenas dímeros de proteína, que consistem em duas proteínas unidas, mas a equipe quer saber a limitação de como as formas das proteínas podem evoluir para formar estruturas hierárquicas de proteínas.
"Antes de fazermos este estudo, Na verdade, eu não esperava que as proteínas pudessem formar dímeros com base apenas na forma, "disse Fengyi Gao, Ph.D. candidato na UM. "Mas agora, descobrimos que isso funciona, e podemos estudar estruturas mais complexas ou até mesmo combinar isso com outras abordagens, como aprendizado de máquina, para ver quais recursos precisamos para habilitar a vinculação correta. "
A equipe espera poder prever a ligação de interfaces proteína-proteína em aglomerados de proteínas ou estruturas de cristalização de proteínas.
“Achamos que podemos adaptar esta abordagem para algo como o rastreamento de drogas no futuro, "Disse Gao." Além disso, esperamos que este modelo baseado em forma possa servir como base para estudar a montagem de proteínas em geral. "