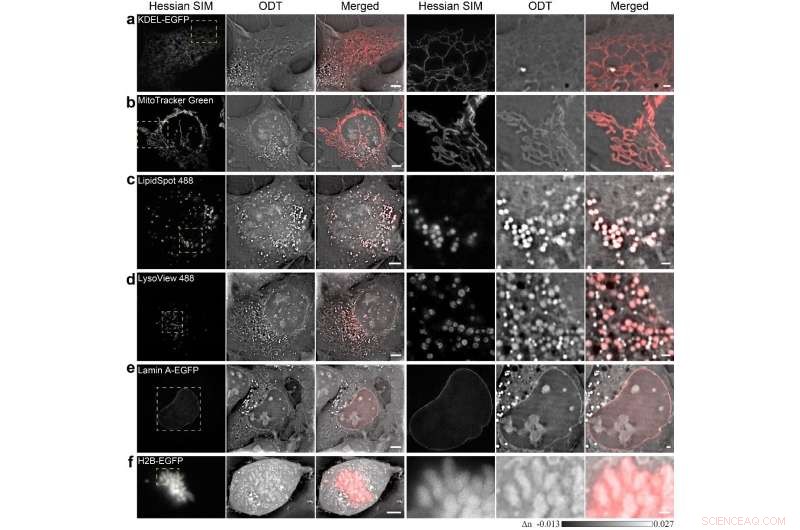
As regiões delimitadas pelas caixas amarelas tracejadas à esquerda são aumentadas à direita. Barras de escala, 5 μm (esquerda) e 1 μm (direita). Crédito:por Dashan Dong, Xiaoshuai Huang, Liuju Li, Heng Mao, Yanquan Mo, Guangyi Zhang, Zhe Zhang, Jiayu Shen, Wei Liu, Zeming Wu, Guanghui Liu, Yanmei Liu, Hong Yang, Qihuang Gong, Kebin Shi, Liangyi Chen
O surgimento da microscopia de fluorescência de superresolução (SR) tem rejuvenescido a busca por novas subestruturas celulares e intermediários dinâmicos. Contudo, limitado pelo amplo espectro de emissão de fluoróforos e fototoxicidade excessiva, A microscopia de fluorescência SR só pode ser usada para destacar um punhado de biomoléculas simultaneamente e é incapaz de fornecer um mapa holístico do ambiente celular e da paisagem.
Em um novo artigo publicado em Ciência leve e aplicações , cientistas do Laboratório Estadual de Física Mesoscópica e Centro de Ciência de Fronteiras para Nano-optoeletrônica, Universidade de Pequim, China, Laboratório Estadual de Biologia de Membrana, Laboratório Chave de Medicina Molecular Cardiometabólica de Pequim, Instituto de Medicina Molecular, Universidade de Pequim, China e colegas desenvolveram tomografia computadorizada de difração assistida por fluorescência SR (SR-FACT), que combina tomografia de difração óptica tridimensional sem rótulo (ODT) com microscopia de iluminação estruturada de Hessian de fluorescência bidimensional. O módulo ODT é capaz de resolver mitocôndrias, gotículas de lipídios, a membrana nuclear, cromossomos, o retículo endoplasmático tubular e os lisossomas. Usando imagens de células vivas correlacionadas de modo duplo por um período prolongado de tempo, eles observaram novas estruturas subcelulares chamadas corpos vacúolos escuros (DBs), a maioria dos quais se originam de regiões perinucleares densamente povoadas, e interagem intensamente com organelas, como a mitocôndria e a membrana nuclear, antes de entrar em colapso na membrana plasmática. Este trabalho demonstra os recursos exclusivos do SR-FACT, o que sugere sua ampla aplicabilidade na biologia celular em geral.
SR-FACT visualiza a paisagem celular e a identidade molecular das células vivas. Um novo algoritmo denominado algoritmo de busca iterativa vetorial (VISA) foi desenvolvido para minimizar erros de reconstrução de imagem 3-D sob esquema de varredura tomográfica de alta velocidade em kHz. Como resultado, SR-FACT pode utilizar simultaneamente uma velocidade máxima de imagem para capturar a dinâmica em células vivas e manter fluxo de fótons suficiente para sensibilidade máxima. No sistema SR-FACT, o módulo ODT atingiu uma resolução lateral de ~ 200 nm a uma velocidade de imagem volumétrica de 0,8 Hz (40 × 40 × 20 m 3 ) Hessian 2-D-SIM, que permite a imagem SR em uma fração da dose de fóton usada pelo SIM convencional, foi utilizado para orientar a interpretação das estruturas observadas pelo módulo ODT. Ao realizar imagens correlacionadas de modo duplo em células COS-7, eles resolveram seis organelas conhecidas sem marcação:o retículo endoplasmático tubular (RE), mitocôndria, endossomos / lisossomos tardios (LEs / LYs), LDs, a membrana nuclear e os cromossomos. Todos esses dados destacam a vantagem única do SR-FACT no estudo do interactome da organela. Além disso, eles também observaram estruturas vacuoladas com pH neutro que continham principalmente líquido no lúmen. A imagem SR de células vivas com lapso de tempo de uma hora em combinação com a análise quantitativa revela as rotas de tráfico não convencionais e os papéis indispensáveis dos vacúolos na organização do interactome organela, tudo isso sugere que eles representam organelas anteriormente não apreciadas.