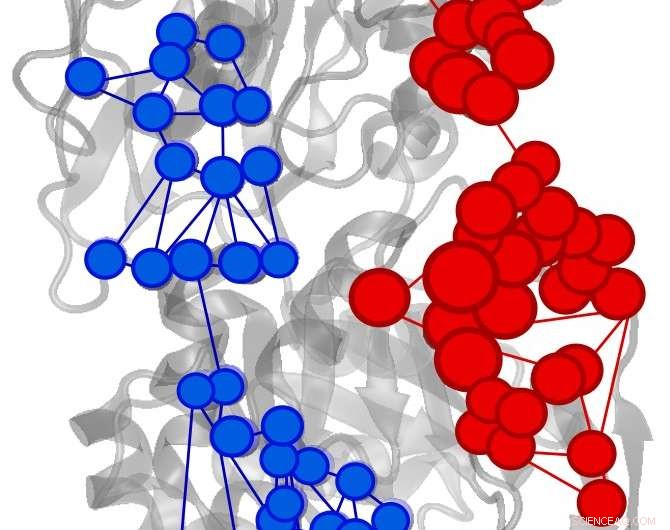
O efetor desencadeou aumento (vermelho) ou diminuição (azul) do fluxo de informações na enzima IGPS. Crédito:Uriel Morzan
Os cientistas de Yale adotaram uma abordagem inovadora para desvendar a complexa estrutura e regulação das enzimas:eles pesquisaram no Google.
Em um novo estudo publicado online esta semana no Proceedings of the National Academy of Sciences , O professor de química Victor Batista e seus colegas usaram o algoritmo do Google PageRank para identificar os aminoácidos-chave na regulação de uma enzima bacteriana essencial para a maioria dos microrganismos.
As enzimas são biomoléculas com a capacidade única de acelerar as reações químicas necessárias à vida. Embora essas reações químicas normalmente ocorram em uma pequena porção da enzima - conhecida como sítio ativo -, a aceleração da reação é geralmente regulada pela ligação de uma molécula em uma parte diferente da enzima. A posição de ligação é conhecida como sítio alostérico.
Apesar de décadas de estudo, ainda é pouco compreendido como a informação é transferida do sítio alostérico para o sítio ativo. Grande parte da dificuldade tem a ver com o grande número de átomos envolvidos e a grande flexibilidade estrutural das enzimas.
A equipe de Yale observou que uma questão semelhante havia sido abordada anos antes no campo da ciência da computação. Pesquisadores do Google estudaram o fluxo de informações na Internet, usando o PageRank para indicar a importância de cada página da web em termos de número e qualidade de links para outros sites da Internet.
“Este problema é completamente análogo à troca de informações entre sites distantes que caracteriza o alosterismo, "disse Uriel Morzan, um associado de pós-doutorado no laboratório de Batista e co-primeiro autor do estudo. "Ao descobrir como o fluxo de informações através de cada átomo muda com a ligação de um ativador alostérico à enzima, é possível encontrar os canais de informação que estão sendo ativados. "
Os pesquisadores de Yale identificaram aminoácidos importantes para o processo alostérico na imidazol glicerol fosfato sintase (IGPS), uma enzima bacteriana encontrada na maioria dos microorganismos.
A pesquisa abre caminho para experimentos adicionais relacionados à atividade IGPS que podem levar ao desenvolvimento de novos antibióticos, pesticidas, e herbicidas.
"É empolgante que os métodos de ciência de dados estejam começando a se infiltrar no campo da química teórica, fornecer novas ferramentas para a compreensão de aspectos fundamentais dos sistemas moleculares catalíticos quando combinados com simulações de dinâmica molecular de última geração e espectroscopia de ressonância magnética nuclear (NMR), "disse Batista, que também é membro do Instituto de Ciências da Energia no Campus Oeste de Yale.
Co-autor J. Patrick Loria, um professor de química e de biofísica molecular e bioquímica de Yale, acrescentou:"É a combinação sinérgica de NMR experimental e ferramentas computacionais que permite uma visão mais profunda da função biológica e demonstra a importância da colaboração entre teóricos e experimentalistas."