 p Imagem de microscópio de força atômica colorida de estruturas de árvore aleatórias automontadas na superfície de matrizes de blocos de DNA. Cada árvore tem um único loop como "raiz". Crédito:Caltech / Grigory Tikhomirov, Philip Petersen e Lulu Qian.
p Imagem de microscópio de força atômica colorida de estruturas de árvore aleatórias automontadas na superfície de matrizes de blocos de DNA. Cada árvore tem um único loop como "raiz". Crédito:Caltech / Grigory Tikhomirov, Philip Petersen e Lulu Qian.
p Muitos sistemas auto-organizados na natureza exploram uma combinação sofisticada de processos determinísticos e aleatórios. Não existem duas árvores exatamente iguais porque o crescimento é aleatório, mas uma sequóia pode ser facilmente distinguida de um jacarandá, pois as duas espécies seguem programas genéticos diferentes. O valor da aleatoriedade em organismos biológicos não é totalmente compreendido, mas tem-se a hipótese de que permite tamanhos menores de genoma - porque nem todos os detalhes devem ser codificados. A aleatoriedade também fornece a variação subjacente à evolução adaptativa. p Em contraste com a biologia, a engenharia raramente tira proveito do poder da aleatoriedade para fabricar estruturas complexas. Agora, um grupo de cientistas da Caltech demonstrou que a aleatoriedade na automontagem molecular pode ser combinada com regras determinísticas para produzir nanoestruturas complexas de DNA.
p O trabalho, feito no laboratório da Professora Assistente de Bioengenharia Lulu Qian, aparece na edição de 28 de novembro da revista
Nature Nanotechnology .
p Os seres vivos usam o DNA para armazenar informações genéticas, mas o DNA também pode ser usado como um bloco de construção químico robusto para a engenharia molecular. As quatro moléculas complementares que compõem o DNA, chamados nucleotídeos, ligam-se apenas de maneiras específicas:A's se ligam a T's, e os G's ligam-se aos C's. Em 2006, Paul Rothemund (BS '94), professor pesquisador de bioengenharia, computação e ciências matemáticas, e sistemas de computação e neurais na Caltech, inventou uma técnica chamada origami de DNA que aproveita a combinação entre longas fitas de nucleotídeos de DNA, dobrando-os em tudo, desde arte em nanoescala a dispositivos de entrega de drogas. As estruturas automontadas formadas por meio de origami de DNA podem ser funcionais por si mesmas ou podem ser usadas como modelos para organizar outras moléculas funcionais, como nanotubos de carbono, proteínas, nanopartículas de metal, e corantes orgânicos - com programação e precisão espacial sem precedentes.
p Usando origami de DNA como um bloco de construção, pesquisadores fizeram nanoestruturas de DNA maiores, como matrizes periódicas de blocos de origami. Contudo, porque o bloco de construção é repetido em todos os lugares, a complexidade dos padrões que podem ser formados nessas estruturas maiores é bastante limitada. Processos de montagem totalmente determinísticos - controlando o design de cada ladrilho individual e sua posição distinta na matriz - podem dar origem a padrões complexos, mas esses processos não aumentam bem. Por outro lado, se apenas processos aleatórios estão envolvidos e os recursos globais da matriz não são controlados por regras de design, é impossível criar padrões complexos com propriedades desejadas sem gerar simultaneamente uma grande fração de moléculas indesejadas que são perdidas. Até o trabalho de Qian e seus colegas, combinar processos determinísticos com processos aleatórios nunca foi sistematicamente explorado para criar nanoestruturas de DNA complexas.
 p Deixou, Ladrilhos truchet têm dois arcos que são rotacionalmente assimétricos. Direito, populares jogos de tabuleiro inspirados nas peças Truchet. Crédito:Cortesia de L. Qian
p Deixou, Ladrilhos truchet têm dois arcos que são rotacionalmente assimétricos. Direito, populares jogos de tabuleiro inspirados nas peças Truchet. Crédito:Cortesia de L. Qian
p "Estávamos procurando princípios de automontagem molecular que englobassem aspectos determinísticos e aleatórios, "diz Qian." Desenvolvemos um conjunto simples de regras que permitem que os blocos de DNA se liguem aleatoriamente, mas apenas em padrões controlados específicos. "
p A abordagem envolve o projeto de padrões em blocos individuais, modulando as proporções de diferentes blocos, e determinar quais ladrilhos podem se ligar durante a automontagem. Isso leva a características emergentes em grande escala com propriedades estatísticas ajustáveis - um fenômeno que os autores chamam de "desordem programável".
p "As estruturas que podemos construir têm aspectos aleatórios de forma programável, "diz Grigory Tikhomirov, um bolsista sênior de pós-doutorado em biologia e engenharia biológica, e autor principal do artigo. "Por exemplo, podemos fazer estruturas com linhas que seguem caminhos aparentemente aleatórios, mas podemos garantir que eles nunca se cruzem e sempre acabem fechando em loops. "
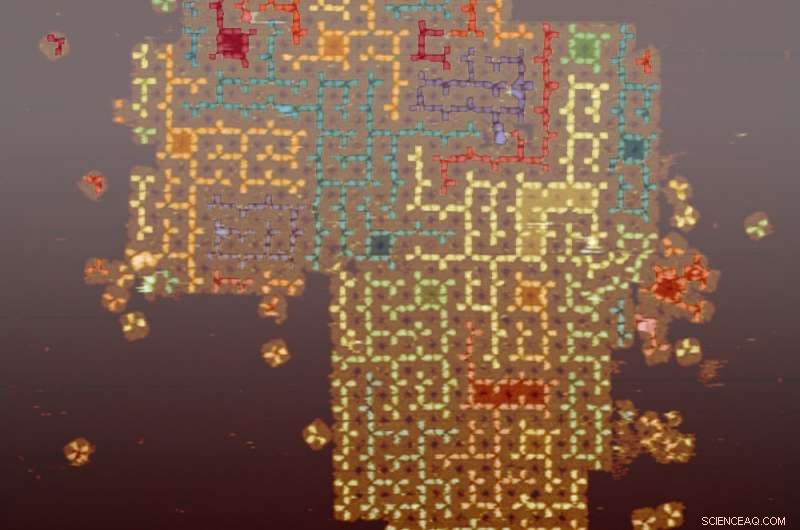
p Além de loops, a equipe escolheu dois outros exemplos, labirintos e árvores, para demonstrar que muitas propriedades não triviais dessas estruturas podem ser controladas por regras locais simples. Eles acharam esses exemplos interessantes porque o loop, Labirinto, e estruturas de árvore existem amplamente na natureza em várias escalas. Por exemplo, pulmões são estruturas de árvore na escala de milímetros a centímetros, e os dendritos neurais são estruturas em árvore na escala de micrômetro a milímetro. As propriedades controladas que eles mostraram incluem as regras de ramificação, as direções de crescimento, a proximidade entre redes adjacentes, e a distribuição do tamanho.
p O grupo foi inicialmente inspirado pelos clássicos azulejos Truchet, que são ladrilhos quadrados com dois arcos de DNA diagonalmente simétricos na superfície. Existem duas orientações rotacionalmente assimétricas do padrão de arco. Permitindo uma escolha aleatória das duas orientações de bloco em cada local na matriz, o padrão continuará pelos blocos vizinhos, tornando-se loops de vários tamanhos ou saindo de uma borda da matriz.
 p Loop auto-montado, Labirinto, e estruturas de árvore na superfície de matrizes de blocos de DNA. Linha superior, labirintos aleatórios com junções de três e quatro vias de distâncias variáveis entre junções adjacentes versus apenas junções de três vias de uma distância fixa entre junções adjacentes. Linha do meio, árvores aleatórias (cada árvore tem um único loop como "raiz") com galhos mais longos de comprimentos variados versus galhos mais curtos de comprimentos fixos. Linha inferior, loops aleatórios com comprimentos ajustáveis e número de cruzamentos. Crédito:L. Qian
p Loop auto-montado, Labirinto, e estruturas de árvore na superfície de matrizes de blocos de DNA. Linha superior, labirintos aleatórios com junções de três e quatro vias de distâncias variáveis entre junções adjacentes versus apenas junções de três vias de uma distância fixa entre junções adjacentes. Linha do meio, árvores aleatórias (cada árvore tem um único loop como "raiz") com galhos mais longos de comprimentos variados versus galhos mais curtos de comprimentos fixos. Linha inferior, loops aleatórios com comprimentos ajustáveis e número de cruzamentos. Crédito:L. Qian
p Para criar matrizes de Truchet em escala molecular, a equipe usou a técnica de origami de DNA para dobrar o DNA em ladrilhos quadrados e, em seguida, projetou as interações entre esses ladrilhos para incentivá-los a se automontarem em grandes matrizes bidimensionais.
p "Porque todas as moléculas se chocam enquanto flutuam em um tubo de ensaio durante o processo de automontagem, as interações devem ser fracas o suficiente para permitir que os ladrilhos se reorganizem e evitar serem presos em quaisquer configurações indesejadas, "diz Philip Petersen, um estudante de pós-graduação no laboratório Qian e co-primeiro autor no artigo. "Por outro lado, as interações devem ser específicas o suficiente para que as interações desejadas sejam sempre preferidas em relação às indesejadas, interações espúrias. "
p Diferentes tipos de padrões globais surgem quando os ladrilhos são marcados com diferentes padrões locais. Por exemplo, se cada ladrilho orientado aleatoriamente carrega um "T" em vez de dois arcos, o padrão global é um labirinto com ramificações e loops, em vez de apenas loops. Se as regras de automontagem restringem a possível orientação relativa dos blocos "T" vizinhos, é possível garantir que, além de uma única "raiz, "os galhos nos labirintos nunca se fecham em loops - produzindo árvores. Para explorar a generalidade desses princípios, A equipe de Qian desenvolveu uma linguagem de programação para peças de origami de DNA aleatórias.
p "Com esta linguagem de programação, o processo de design começa com uma descrição de alto nível dos blocos e matrizes, que pode ser traduzido automaticamente em diagramas de matriz abstrata e simulações numéricas, em seguida, passa para o design de ladrilhos de origami de DNA, incluindo como os ladrilhos interagem uns com os outros em suas bordas. Finalmente, nós projetamos sequências de DNA, "Qian diz." Com essas sequências de DNA, é simples para os pesquisadores ordenarem as fitas de DNA, misture-os em um tubo de ensaio, espere as moléculas se automontarem nas estruturas projetadas durante a noite, e obter imagens das estruturas usando um microscópio de força atômica. "
p O método de desordem programável do grupo tem diversas aplicações futuras. Por exemplo, poderia ser usado para construir ambientes de teste complexos para robôs moleculares cada vez mais sofisticados - máquinas em nanoescala baseadas em DNA que podem se mover em uma superfície, pegar ou soltar proteínas ou outros tipos de moléculas como cargas, e tomar decisões sobre navegação e ações.
p "As aplicações potenciais são muito mais amplas, "Qian acrescenta. Desde a década de 1990, cadeias unidimensionais aleatórias de polímeros têm sido usadas para revolucionar a síntese química e de materiais, entrega de drogas, e química de ácido nucléico, criando vastas bibliotecas combinatórias de moléculas candidatas e, em seguida, selecionando ou desenvolvendo as melhores no laboratório. "Nosso trabalho estende o mesmo princípio para redes bidimensionais de moléculas e agora cria novas oportunidades para a fabricação de dispositivos moleculares mais complexos organizados por nanoestruturas de DNA, " ela diz.
p O artigo é intitulado "Desordem programável em camadas aleatórias de DNA".


