 p Trecho do filme de lapso de tempo de detecção de miRNA em 1 hora usando o ensaio miRacles. Crédito:Science Advances, doi:10.1126 / sciadv.aau9443
p Trecho do filme de lapso de tempo de detecção de miRNA em 1 hora usando o ensaio miRacles. Crédito:Science Advances, doi:10.1126 / sciadv.aau9443
p MicroRNAs (miRNAs) são pequenos RNAs reguladores não codificadores que podem reprimir a expressão gênica pós-transcricionalmente e, portanto, são cada vez mais usados como biomarcadores de doenças. A detecção de miRNAs pode ser árdua e cara, pois requerem amplificação, rotulagem e sondas radioativas. Em um relatório recente publicado em
Avanços da Ciência , Arun Richard Chandrasekaran e colegas de trabalho do Instituto de RNA e do Departamento de Ciências Biológicas, na Universidade de Albany, Universidade Estadual de Nova York, relatado em uma única etapa, ensaio de detecção de microRNA não enzimático usando nanointerruptores de DNA conformacionalmente não responsivos. p Os cientistas chamaram o ensaio de 'miRacles, 'para abreviar' looping condicional ativado por micro-RNA de interruptores projetados '. O ensaio demonstrou subattomole e especificidade de nucleotídeo único usando uma leitura de eletroforese em gel de agarose. Nos experimentos, eles detectaram microRNAs celulares de extrações de RNA obtidas de músculos em diferenciação na escala de nanogramas. Os cientistas apresentaram uma configuração experimental econômica para detectar miRNAs em um período de horas para fornecer uma alternativa atraente para os métodos existentes de reação em cadeia da polimerase quantitativa (qPCR) e Northern blotting para quantificar os materiais genéticos regulatórios.
p miRNAs podem regular muitos processos biológicos durante o desenvolvimento fisiológico normal e doenças, afetando a proliferação celular, diferenciação e apoptose in vivo. A expressão de miRNAs pode ser quantificada nos tecidos, células e fluidos corporais como biomarcadores estáveis para eventos celulares e diagnóstico de doenças, destacando a importância de sua detecção. No entanto, A detecção de miRNA é um desafio devido à baixa abundância, tamanho pequeno e semelhanças na sequência. As biomoléculas compreendem aproximadamente 0,01 por cento do conteúdo total de RNA e as cópias individuais de mRNA por faixa de células podem variar amplamente. Adicionalmente, miRNAs em uma família podem diferir por um único nucleotídeo, enquanto miRNAs específicos podem ser regulados durante a doença e função celular regular. Como resultado, as estratégias de detecção de miRNA requerem alta especificidade e a capacidade de identificar corretamente algumas moléculas de uma amostra abundante com moléculas de RNA predominantes.
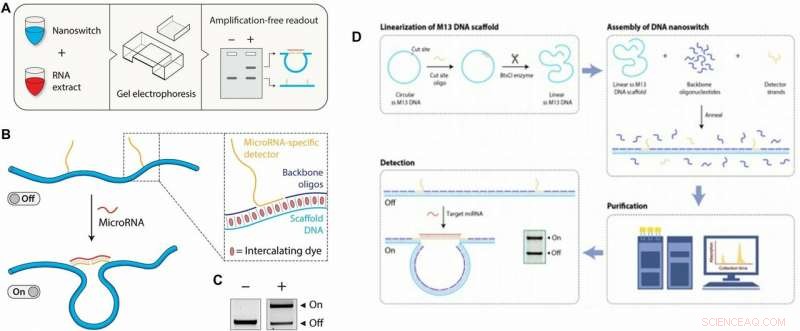 p Esquerda:Conceito e fluxo de trabalho do ensaio miRacles. (A) Fluxo de trabalho do ensaio miRacles:nanointerruptores de DNA personalizados são misturados com a amostra miRNA alvo, incubado, e correr em um gel de agarose para detecção. (B) Os nanointerruptores de DNA passam por uma mudança conformacional de um estado "desligado" linear para um estado "ligado" em loop quando ligados a um miRNA alvo. Detalhe:o nanoswitch é composto por um andaime M13 de fita simples, oligonucleotídeos de base, e extensões de fita simples (detectores) complementares ao miRNA alvo. Corantes intercalados intrínsecos ao processo de eletroforese fornecem o sinal para visualizar os nanointerruptores. (C) As duas conformações são resolvidas em um gel de agarose padrão. À direita:Exame aprofundado do processo de construção do nanointerruptor de DNA. (D) Linearização:A preparação de um arcabouço M13 de fita simples linear prossegue por emparelhamento de um oligonucleotídeo para permitir a clivagem no local de fita dupla por uma enzima de restrição (BtsCI neste caso). Montagem:a montagem do nanointerruptor de DNA é realizada pela mistura de um excesso molar de 10x de oligonucleotídeos de backbone e fitas detectoras (específicas para o alvo) para o M13 de fita simples e aplicação de uma rampa de resfriamento de 90˚C a 25˚C a 1˚C / min . Purificação:Para remover o excesso de oligonucleotídeos, um método de cromatografia líquida é usado. Detecção:Para usar os nanointerruptores, eles são misturados com uma solução contendo alvos de microRNA e executados em um gel para resolver os estados ligado e desligado, conforme mostrado na inserção. Crédito:Science Advances, doi:10.1126 / sciadv.aau9443
p Esquerda:Conceito e fluxo de trabalho do ensaio miRacles. (A) Fluxo de trabalho do ensaio miRacles:nanointerruptores de DNA personalizados são misturados com a amostra miRNA alvo, incubado, e correr em um gel de agarose para detecção. (B) Os nanointerruptores de DNA passam por uma mudança conformacional de um estado "desligado" linear para um estado "ligado" em loop quando ligados a um miRNA alvo. Detalhe:o nanoswitch é composto por um andaime M13 de fita simples, oligonucleotídeos de base, e extensões de fita simples (detectores) complementares ao miRNA alvo. Corantes intercalados intrínsecos ao processo de eletroforese fornecem o sinal para visualizar os nanointerruptores. (C) As duas conformações são resolvidas em um gel de agarose padrão. À direita:Exame aprofundado do processo de construção do nanointerruptor de DNA. (D) Linearização:A preparação de um arcabouço M13 de fita simples linear prossegue por emparelhamento de um oligonucleotídeo para permitir a clivagem no local de fita dupla por uma enzima de restrição (BtsCI neste caso). Montagem:a montagem do nanointerruptor de DNA é realizada pela mistura de um excesso molar de 10x de oligonucleotídeos de backbone e fitas detectoras (específicas para o alvo) para o M13 de fita simples e aplicação de uma rampa de resfriamento de 90˚C a 25˚C a 1˚C / min . Purificação:Para remover o excesso de oligonucleotídeos, um método de cromatografia líquida é usado. Detecção:Para usar os nanointerruptores, eles são misturados com uma solução contendo alvos de microRNA e executados em um gel para resolver os estados ligado e desligado, conforme mostrado na inserção. Crédito:Science Advances, doi:10.1126 / sciadv.aau9443
p Os métodos tradicionais para detecção de miRNA incluem Northern blotting, reação em cadeia da polimerase de transcrição reversa quantitativa (qRT-PCR), sequenciamento de última geração e hibridização baseada em microarray para separar o sinal do miRNA do ruído. Dos métodos especificados, O Northern blotting pode identificar diretamente miRNAs nativos, enquanto outros contam com métodos de rotulagem adicionais ou amplificação passo a passo, adicionando ao custo, complexidade e desempenho da detecção. Por exemplo, nanoestruturas de DNA inovadoras podem ser usadas para detecção de miRNA, onde vários grupos de pesquisa já combinaram nanoestruturas com nanopartículas, reações em cadeia de hibridização e nanofolhas de dichalcogeneto de metal de transição para permitir o processo.
p No presente trabalho, Chandrasekaran et al. desenvolveu um dispositivo baseado em DNA relativamente simples para resolver um desafio biomédico complexo. No ensaio miRacles, os cientistas usaram um 'reagente inteligente' composto de nanointerruptores de DNA racionalmente projetados para detecção simples e econômica de miRNA nativo, sem o uso de equipamento especializado no laboratório. Os nanointerruptores de DNA foram originalmente projetados como ferramentas para experimentos de biofísica de uma única molécula e, posteriormente, reconhecidos por sua capacidade de detectar e quantificar as interações biomoleculares usando eletroforese em gel. Os esforços de pesquisa colaborativa anteriores da mesma equipe de pesquisa focaram na detecção molecular para quantificar os níveis de proteína e detectar sequências de DNA sintético como uma prova de conceito.
p O presente trabalho expandiu estudos preliminares e conceitos para produzir detecção e quantificação de miRNA multiplexados prontos para o usuário. Os cientistas analisaram nanogramas de extratos de RNA celular em um curto espaço de tempo usando uma configuração experimental construída com materiais comuns de laboratório. Eles projetaram o nanoswitch de DNA como um duplex linear que formou um loop na presença da molécula de miRNA alvo. Para construir o nanoswitch, Chandrasekaran et al. usou a abordagem de origami de DNA por hibridização de oligonucleotídeos curtos complementares a um andaime de DNA de fita simples.
p Eles projetaram dois fios "detectores" distantes com saliências complementares a diferentes segmentos do miRNA alvo. Quando o miRNA é reconhecido e ligado ao construto, o switch foi reconfigurado do estado "desligado" linear para o estado "ligado" em loop. Eles quantificaram os dois estados usando eletroforese em gel de agarose padrão para detectar o sinal proveniente do nanoswitch em loop. O sinal foi amplificado apenas por um único miRNA de interesse, os resultados foram comparados favoravelmente com a técnica de transferência de energia de ressonância de fluorescência (FRET).
 p Validação do ensaio miRacles. (A) Especificidade dos nanointerruptores de DNA com detectores projetados para let-7b. A incompatibilidade de até 1 nt entre os detectores e o miRNA alvo elimina o sinal. (B) Limite de detecção do ensaio. NC, controle negativo. AU, unidades arbitrárias. (C) Decurso do tempo do ensaio para um alvo de baixa concentração. (D) Faixa dinâmica do ensaio em diferentes tempos de reação. Crédito:Science Advances, doi:10.1126 / sciadv.aau9443.
p Validação do ensaio miRacles. (A) Especificidade dos nanointerruptores de DNA com detectores projetados para let-7b. A incompatibilidade de até 1 nt entre os detectores e o miRNA alvo elimina o sinal. (B) Limite de detecção do ensaio. NC, controle negativo. AU, unidades arbitrárias. (C) Decurso do tempo do ensaio para um alvo de baixa concentração. (D) Faixa dinâmica do ensaio em diferentes tempos de reação. Crédito:Science Advances, doi:10.1126 / sciadv.aau9443.
p Para validar o conceito, a equipe de pesquisadores escolheu um miRNA alvo let-7b devido à sua família altamente conservada de mais de uma dúzia de miRNAs relacionados que variavam por um ou mais nucleotídeos. Esses miRNAs foram adequados devido ao seu papel crítico nas funções biológicas e desregulação em várias doenças humanas. Para eliminar a interferência e o ruído entre o nanoswitch e o alvo, o que levou à redução da intensidade do sinal em comparação com uma combinação perfeita, os cientistas redesenharam racionalmente os nanointerruptores. Para alcançar a especificidade perfeita, eles desestabilizaram a interação do lado que continha a incompatibilidade. Os resultados do estudo ilustraram a alta especificidade do ensaio assim desenvolvido, fornecendo uma resposta a um desafio chave na detecção de miRNA que culminou com uma alta relação sinal / ruído.
p A baixa abundância de miRNAs também exigiu alta sensibilidade de detecção, que os cientistas conseguiram otimizando o protocolo. Eles então realizaram experimentos semelhantes para duas outras variantes de miRNAs (miR-15 a e miR-206), resultando em níveis de detecção de subattomole para single-attomole. Por exemplo, para uma baixa concentração de uma amostra alvo (6 pM), o sinal aumentou por 4 horas, com poucas mudanças além desse período.
p
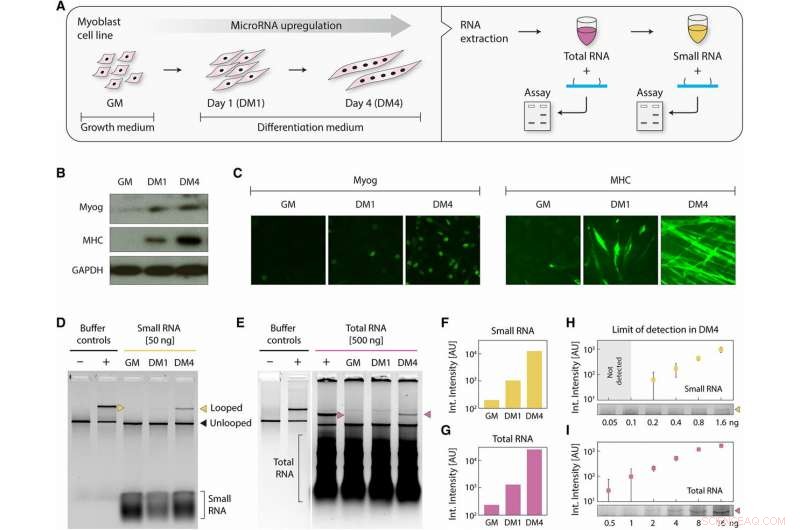 p Detecção de miRNA de células de mioblastos em diferenciação. (A) Esquema mostrando células mioblásticas, colhido durante o crescimento em GM e nos dias de diferenciação 1 e 4, processado para produzir frações de RNA totais e pequenas. Um marcador precoce de diferenciação miogênica, Myog, e um marcador de diferenciação miogênica tardia, MHC, foram medidos por (B) Western blotting e (C) por imunocitoquímica para confirmar a diferenciação. Myog e MHC foram regulados para cima em DM1 e DM4. GAPDH (gliceraldeído-3-fosfato desidrogenase) serviu como um controle em (B). (D) Chandrasekaran et al. detectou miR-206 nas amostras diferenciadas com 50 ng de RNAs pequenos e (E) com 500 ng de RNAs totais. A quantificação das intensidades do gel mostra uma regulação ascendente acentuada e progressiva durante a diferenciação, semelhante em ambas as amostras de (F) RNA pequeno e (G) RNA total. A partir de amostras DM4, notamos a detecção de tão pouco quanto (H) 200 pg de RNAs pequenos e (I) 500 pg de RNAs totais. Crédito:Science Advances, doi:10.1126 / sciadv.aau9443.
p Detecção de miRNA de células de mioblastos em diferenciação. (A) Esquema mostrando células mioblásticas, colhido durante o crescimento em GM e nos dias de diferenciação 1 e 4, processado para produzir frações de RNA totais e pequenas. Um marcador precoce de diferenciação miogênica, Myog, e um marcador de diferenciação miogênica tardia, MHC, foram medidos por (B) Western blotting e (C) por imunocitoquímica para confirmar a diferenciação. Myog e MHC foram regulados para cima em DM1 e DM4. GAPDH (gliceraldeído-3-fosfato desidrogenase) serviu como um controle em (B). (D) Chandrasekaran et al. detectou miR-206 nas amostras diferenciadas com 50 ng de RNAs pequenos e (E) com 500 ng de RNAs totais. A quantificação das intensidades do gel mostra uma regulação ascendente acentuada e progressiva durante a diferenciação, semelhante em ambas as amostras de (F) RNA pequeno e (G) RNA total. A partir de amostras DM4, notamos a detecção de tão pouco quanto (H) 200 pg de RNAs pequenos e (I) 500 pg de RNAs totais. Crédito:Science Advances, doi:10.1126 / sciadv.aau9443.
miRNAs também são tipicamente estudados em culturas de células, amostras de tecido e fluidos corporais e prontamente processados para coletar RNAs totais na escala do micrograma, junto com uma subfração de pequeno RNA. Por esta, Chandrasekaran et al. usaram células de mioblastos como um sistema modelo substituto de diferenciação muscular, com miR-206 como o alvo principal. Durante a cultura de células, eles identificaram miRNAs na diferenciação de músculos esqueléticos, onde a molécula miR-206 foi significativamente regulada positivamente durante a diferenciação tardia. Quando os cientistas coletaram as células musculares indiferenciadas e diferenciadas para extrair pequenos RNAs, eles confirmaram a diferenciação celular usando Western blots e conduziram imunocitoquímica para marcadores de diferenciação precoce e tardia miogenina (Myog) e cadeia pesada de miosina (MHC), respectivamente. Uma vez que miRNAs individuais compõem o nível de partes por milhão em massa de RNAs totais, detectar miRNAs é tão desafiador quanto encontrar uma agulha em um palheiro. Para validar o ensaio para detecção biológica, Portanto, Chandrasekaran et al. começou com a pequena subfração de RNA menos complexa. Ao usar o nanoswitch alvo miR-206, os cientistas observaram um aumento progressivo na expressão do miR-206 em pequenos RNAs entre amostras indiferenciadas e amostras diferenciadas por 1-4 dias.
p Uma vez que vários mRNAs também podem alterar sua expressão durante diferentes estágios celulares ou de doença, o fenômeno exigia recursos adicionais de detecção. Para fazer isso na configuração experimental, Chandrasekaran et al. usou a programabilidade de nanointerruptores e desenvolveu um sistema de multiplexação capaz de detectar vários miRNAs da mesma amostra. Eles colocaram as fitas do detector em locais desejados da estrutura de DNA, resultando em loops de tamanhos diferentes quando ligados ao miRNA alvo. O tamanho do loop do nanointerruptor determinou assim a migração do gel, resultando em uma banda única no gel para detecção precisa. Para o experimento, os cientistas escolheram quatro miRNAs presentes nas células musculares; miR-206, miR-125b, miR-24 e miR-133-3p e um miRNA de controle negativo; miR-39 específico para a espécie
Caenorhabditis elegans.
p Dentro de 50 ng de pequenos RNAs, os cientistas detectaram os quatro miRNAs em vários níveis de expressão, enquanto confirma nenhuma detecção com o controle negativo. A estratégia de multiplexação permitiu aos cientistas comparar diretamente os níveis de miRNA em uma única amostra, sem rotulagem ou amplificação. No total, o trabalho forneceu um passo adicional na direção de expandir o rendimento do ensaio miRacles. A capacidade pode ser expandida para acomodar mais miRNAs por switch também.
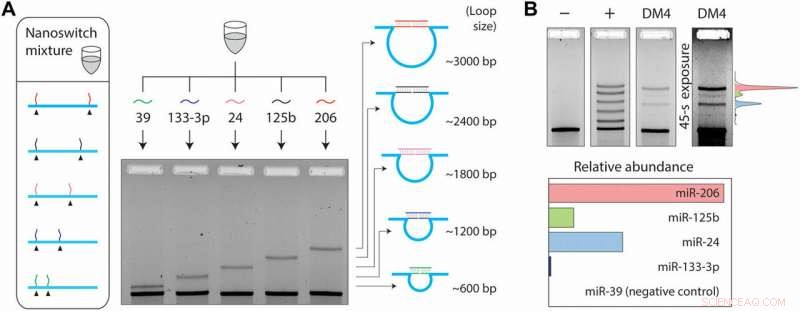 p Configuração de multiplexação de cinco canais. (A) A multiplexação permite a detecção de diferentes miRNAs com diferentes tamanhos de loop. (B) Uma mistura de nanoswitch multiplexada mostra cinco bandas com intensidade semelhante em um controle positivo que consiste em todos os cinco miRNAs alvo. Em 50 ng de pequenos RNAs de DM4, quatro miRNAs diferentes são detectados em vários níveis de expressão, com miR-39 (um miRNA específico de C. elegans) não sendo detectado.
p Configuração de multiplexação de cinco canais. (A) A multiplexação permite a detecção de diferentes miRNAs com diferentes tamanhos de loop. (B) Uma mistura de nanoswitch multiplexada mostra cinco bandas com intensidade semelhante em um controle positivo que consiste em todos os cinco miRNAs alvo. Em 50 ng de pequenos RNAs de DM4, quatro miRNAs diferentes são detectados em vários níveis de expressão, com miR-39 (um miRNA específico de C. elegans) não sendo detectado.
p Desta maneira, Chandrasekaran et al. substancialmente avançou de sua detecção de prova de conceito preliminar de sequências de DNA sintéticas; para estabelecer, caracterizar e otimizar um ensaio de detecção de miRNA pronto para uso com extratos biológicos. O trabalho demonstrado foi um primeiro exemplo de uso de nanointerruptores de DNA para detectar miRNAs de uma amostra biológica verdadeira. Enquanto o desempenho do ensaio de miRNA foi competitivo em comparação com outras técnicas comumente usadas, a seletividade de 1 nucleotídeo visto no presente trabalho foi difícil de realizar com os métodos existentes. A sensibilidade dos miRacles também superou o Northern blotting e os microarrays. O ensaio pode medir miRNAs sem a necessidade de amplificação, com protocolos mais simples e sem o erro adicional de processamento de amostra extra. O protocolo simplesmente misturou os nanointerruptores com o líquido de amostra para eletroforese em gel, para produzir resultados de alta qualidade no laboratório. O trabalho de pesquisa é potencialmente transferível de amostras biológicas para clínicas para diagnosticar e monitorar doenças.
p Mais importante, o presente trabalho alinha-se com o conceito mais amplo de ciência econômica; uma visão promissora de ciência econômica que já produziu soluções de baixo custo para técnicas de centrifugação de sangue e purificação de água em engenharia biomédica. Chandrasekaran et al. pretendem continuar a contribuir para a tendência emergente na ciência, interrompendo a relação custo / desempenho existente para fornecer acesso em larga escala a métodos simples de detecção de miRNA com reagentes inteligentes de leitura e mistura. p © 2019 Science X Network




